Fronteras en Microbiología
Introducción
En la última década, una especie emergente de Candida nosocomial ha estado causando casos graves de candidemia y otros tipos de infecciones fúngicas. Se han observado brotes del patógeno responsable, concretamente Candida auris, en todo el mundo (véase la Figura 1A) (Lockhart et al., 2017).
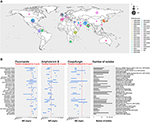
Figura 1. (A) Mapa que representa el número de aislados por brote desde 1996. El primer año del brote está coloreado en marrón y el último en rosa. El tamaño del círculo es proporcional al número de aislados notificados para cada brote. El estudio de brotes se realizó recuperando los datos de tres revisiones principales (Osei Sekyere, 2018; Jackson et al., 2019; Kenters et al., 2019) y se complementó con una búsqueda en pubmed utilizando la consulta «Candida» AND «auris» AND «outbreak» como palabras clave. Los resultados fueron limitados en el tiempo, entre el 21/07/2017, el último estudio de la revisión sistemática de Osei Sekyere y el 31/08/2019, cuando se realizó la búsqueda bibliográfica. Estudios no presentes en la revisión de Osei Sekyere: (Biswal et al., 2017; Abdalhamid et al., 2018; Adams et al., 2018; Belkin et al., 2018; Chow et al., 2018; Desoubeaux et al., 2018; Lesho et al., 2018; Mayor, 2018; Parra-Giraldo et al., 2018; Ruiz-Gaitán et al, 2018, 2019a,b; Sexton et al., 2018; Adam et al., 2019; Barantsevich et al., 2019; Escandón et al., 2019; Leach et al., 2019; O’Connor et al., 2019; Park et al., 2019; Sana et al., 2019; Sayeed et al., 2019). (B) Rangos de CIM para tres antifúngicos medidos en aislados de C. auris de diferentes brotes en todo el mundo. La línea roja punteada representa el punto de ruptura tentativo propuesto por los CDC. IPSV es un acrónimo de «India, Pakistán, Sudáfrica y Venezuela», ya que las cepas de estos países fueron analizadas en el mismo estudio. El estudio de los brotes se realizó de forma idéntica a (A). Sólo se representaron los brotes con >1 aislado. Los estudios citados por Osei Sekyere están señalados con y los estudios citados por Kenters et al. con .
La secuenciación del genoma completo determinó la presencia de cuatro clados específicos dentro de la especie C. auris: Asia oriental, Asia meridional, Sudáfrica y Sudamérica. Todos los clados se caracterizan por polimorfismos de un solo nucleótido (SNP) distintos, lo que pone de manifiesto la emergencia independiente y mundial de este patógeno (Lockhart et al., 2017).
Estudios comparativos en diferentes modelos animales (murinos y larvas de Galleria mellonella) mostraron niveles de virulencia dependientes de la cepa para C. auris. En la mayoría de los casos, las cepas de C.auris resultaron ser menos virulentas que las de C. albicans (Borman et al., 2016; Ben-Ami et al., 2017; Fakhim et al., 2018; Wang et al., 2018). Sin embargo, los casos reportados de brotes de C. auris se relacionaron con una alta tasa de mortalidad, principalmente en pacientes inmunocomprometidos. Otras condiciones como la diabetes, las enfermedades cardiovasculares y/o pulmonares, la sepsis o un tratamiento previo con antibióticos parecían ser también factores de riesgo importantes (Osei Sekyere, 2018).
Excepcionalmente para una levadura, C. auris se ha propagado con éxito en entornos nosocomiales. Por ejemplo, en un hospital del Reino Unido, un solo paciente colonizado por C. auris dio lugar a posteriores identificaciones de otros casos entre otros pacientes hospitalizados. El patógeno también se detectó en el personal sanitario incluso varios meses después de su primer aislamiento, lo que sugiere una eficiente transmisión entre humanos. Además, se encontraron rastros de C. auris en los lados de las camas, alféizares, monitores y otros equipos en todo el hospital, lo que demuestra que C. auris es capaz de persistir en las superficies nosocomiales (Schelenz et al., 2016), en contraste con otras especies de Candida. La investigación de un brote en otro hospital del Reino Unido informó de que el uso de equipos reutilizables, como las sondas de temperatura axilar, fue la principal causa de transmisión a los pacientes (Eyre et al., 2018), lo que ilustra aún más su capacidad de sobrevivir en las superficies.
La capacidad de sobrevivir en entornos nosocomiales podría deberse a la capacidad de C. auris de formar biopelículas; una forma de crecimiento en la que las células se disponen juntas en microcolonias protegidas por una matriz de glucano. Por convención, las células encerradas en la matriz se denominan sésiles, y las que están fuera de ella, planctónicas. Se ha demostrado que las células de C. auris protegidas por esta estructura son difíciles de eliminar, incluso con procedimientos de desinfección de alto nivel (Kean et al., 2018b).
Además de permitir potencialmente una mayor supervivencia en superficies inertes, la capacidad de C. auris para formar biofilms también puede ser un factor importante de su resistencia a los antifúngicos sistémicos utilizados en el tratamiento de los pacientes. De hecho, el hecho de que se hayan aislado células formadoras de biofilms de heridas y puntas de catéteres indica que algunas células de C. auris podrían sobrevivir en forma de biofilm en los pacientes (Borman et al., 2016) y se demostró que las células sésiles presentaban una susceptibilidad reducida para varios antifúngicos. (Sherry et al., 2017). Por último, también se ha demostrado que el biofilm es un importante factor de virulencia, ya que las cepas de C. auris que forman biofilm están relacionadas con una mayor morbilidad y mortalidad (Borman et al., 2016).
Con esta revisión pretendemos destacar los mecanismos de resistencia molecular adquiridos por C. auris, que llevaron a su extraordinaria supervivencia en el entorno nosocomial y en los pacientes hospitalizados.
Susceptibilidad antifúngica de C. auris
Después de 10 años de registro de la concentración inhibitoria mínima (CIM) para los antifúngicos disponibles, los CDC publicaron recientemente una tentativa de puntos de ruptura para definir mejor la susceptibilidad antifúngica de C. auris1. Se puso de manifiesto que C. auris tiene unas CIM excepcionalmente altas para las cuatro clases principales de antifúngicos: azoles, equinocandinas, polienos (Figura 1B) y análogos de nucleósidos. En este capítulo, examinaremos en detalle dos factores que han demostrado desempeñar un papel en esta menor susceptibilidad a los tratamientos antifúngicos: los mecanismos de resistencia molecular y los mecanismos de resistencia asociados al biofilm.
2.1. Mecanismos de resistencia molecular
Definimos aquí la resistencia molecular como las formas en que las células individuales pueden disminuir su sensibilidad a los antifúngicos debido a modificaciones genéticas que conducen a la modificación de la diana del fármaco, a la sobreexpresión de las bombas de eflujo o a modificaciones del metabolismo (véase para su revisión: Pemán et al., 2009; Sanglard et al., 2009; Krishnasamy et al., 2018). Esta sección recopila, para cada clase de antifúngico, los principales mecanismos moleculares de resistencia encontrados en C. auris hasta el momento. Además, la Tabla 1 resume para cada fármaco, el modo de acción, los mecanismos de resistencia relacionados descritos en Candida no auris y los mecanismos de resistencia descubiertos en C. auris, hasta el momento.

Tabla 1. Farmacodinámica de las cuatro clases principales de antifúngicos y mecanismos de resistencia vinculados en Candida spp. en general y en C. auris específicamente.
2.1.1. Mecanismos de resistencia a los azoles
2.1.1.1. Sobreexpresión de bombas de eflujo MFS y ABC
Las bombas de eflujo son proteínas que transportan componentes a través de la membrana celular. Algunas de ellas pueden bombear fármacos al exterior de la célula, disminuyendo su concentración y efecto en la misma. Existen dos grandes familias de bombas de eflujo implicadas en la resistencia a los antifúngicos: Los transportadores de casete de unión a ATP (ABC) y los de la superfamilia de facilitadores principales (MFS). La sobreexpresión de las bombas de eflujo es uno de los principales mecanismos de resistencia a los azoles en las especies patógenas de Candida (Schuetzer-Muehlbauer et al., 2003; Morschhäuser et al., 2007).
Las cepas indias de C. auris secuenciadas en dos estudios diferentes presentan numerosos genes ortólogos a los transportadores ABC y MFS de C. albicans (Chatterjee et al., 2015; Sharma et al., 2016). Curiosamente, las cepas de C. auris que se aislaron en Israel mostraron una actividad de transportador ABC intrínsecamente alta en comparación con C. glabrata y C. haemulonii, midiendo el eflujo de un sustrato fluorescente (Rodamina 6G) (Ben-Ami et al., 2017).
En C. albicans, CDR1 es un gen que codifica para una bomba de eflujo ABC, conocida por su papel en la resistencia a los azoles (Sanglard et al., 1995). Se ha encontrado un gen homólogo a CDR1 en C. auris. (Rybak et al., 2019). El mismo estudio también demostró que la deleción de este gen podía aumentar la susceptibilidad de las cepas resistentes de 64 a 128 veces (Rybak et al., 2019).
2.1.1.2. Mutaciones puntuales en ERG11
El ergoesterol es un componente de membrana clave en los hongos. En Candida spp., su biosíntesis está mediada, entre otras, por la enzima Lanosterol 14-alfa-demetilasa (LD), codificada por el gen ERG11 que convierte el lanosterol en ergosterol. La LD es el principal objetivo de los azoles, antifúngicos que inhiben la función de la enzima y detienen eficazmente la biosíntesis de ergosterol, lo que perjudica la integridad de la membrana (Sanglard et al., 1998).
Se ha demostrado que las mutaciones puntuales en ERG11 reducen la sensibilidad a los azoles en Candida spp, particularmente en tres regiones «calientes» localizadas entre los aminoácidos 105-165, 266-287 y 405-488 (Vandeputte et al., 2012).
Interesantemente, después de haber recuperado las secuencias de aminoácidos de 44 aislados de C. auris de la India, se encontraron 15 mutaciones sin sentido cuando se alinearon con la secuencia ERG11 de tipo salvaje de C. albicans (Chowdhary et al., 2018). Cinco de estas mutaciones ya estaban asociadas a la resistencia a los azoles en C. albicans. Entre estas variantes, dos se encontraron en cada cepa resistente analizada en este estudio, a saber, Y132F o K143R (Chowdhary et al., 2018). También se puede observar que estas mutaciones se encuentran en el primer «punto caliente» discutido anteriormente.
Un cribado de variantes en el gen ERG11 de cepas de C. auris de Columbia identificó las mismas sustituciones Y132F y K143R. Tras la expresión heteróloga de estas dos mutaciones en S. cerivisiae, las CMI para los azoles presentaron un aumento del doble en comparación con las cepas de S. cerevisiae que expresaban el gen ERG11 de tipo salvaje de C. auris (Healey et al., 2018).
2.1.1.3. Sobreexpresión de ERG11
En C. albicans, una sobreexpresión de ERG11 se ha relacionado con una resistencia también al tratamiento con azoles.El aumento de la producción de LD desborda la capacidad del antifúngico para inhibir la actividad de la proteína, dando lugar a una proteína activa a pesar del tratamiento farmacológico (López-Ribot et al, 1998).
En C. auris, los experimentos de PCR en tiempo real mostraron que en ausencia de fluconazol, no había diferencia en la expresión de ERG11 entre las cepas susceptibles y resistentes al fluconazol (Chowdhary et al., 2018). En presencia del antifúngico, la expresión de ERG11 aumenta en las cepas resistentes en comparación con un control sin fluconazol (Chowdhary et al., 2018). Sin embargo, el efecto de este fármaco sobre la expresión de ERG11 no se probó en cepas susceptibles. Por lo tanto, no se ha demostrado que el aumento condicional de la expresión de ERG11 en presencia de fluconazol sea específico de las cepas resistentes, hasta ahora.
2.1.2. Mecanismos de resistencia a las equinocandinas
El beta(1,3)D-glucano es un componente clave de la pared celular fúngica y está codificado por los genes FKS1 y FKS2, dos subunidades de la beta(1,3)D-glucano sintasa. Las equinocandinas inhiben esta enzima, disminuyendo la cantidad de glucanos en la pared celular (Martins et al., 2011).
En C. albicans y otras especies de Candida no auris, se encontraron varias mutaciones que conducen a la resistencia a las equinocandinas en las mismas dos regiones de FKS1 y FKS2. Por ello, se denominaron «puntos calientes» 1 y 2 (HS1 y HS2). En el gen FKS1 de C. albicans estos «puntos calientes» se localizan entre los aminoácidos 641-649 y 1.345-1.365 (Park et al., 2005). La secuenciación de las correspondientes regiones de puntos calientes de 38 cepas de C. auris condujo al descubrimiento de una sustitución de aminoácidos S639F que se correlaciona con la resistencia a la pan-echinocandina: estaba presente en las cuatro cepas resistentes y ausente en las 34 susceptibles. Esta posición en C. auris FKS1 está alineada con la posición 645 (también una serina) en C. albicans (Chowdhary et al., 2018). Curiosamente, esta mutación se encuentra en la región que se alinea con la HS1 de C. albicans FKS1. Otros estudios observaron diferentes mutaciones en la misma ubicación en cepas de C. auris resistentes a la equinocandina: S639Y y S639P (Rhodes et al., 2018). Se confirmó que esta última causa resistencia a las equinocandinas in vivo en un modelo de ratón (Kordalewska et al., 2018).
FKS2 también se encontró en una sola copia en el genoma de C. auris (Sharma et al., 2016), pero no se encontró ninguna mutación asociada a la resistencia a las equinocandinas en este gen hasta donde sabemos.
2.1.3. Mecanismos de resistencia a polienos
Como se muestra en la Figura 1B, varias cepas de C. auris son resistentes a la anfotericina B (AMB). En las especies de Candida, se han destacado las modificaciones de la composición de esteroles de la membrana como mecanismo de resistencia (Haynes et al., 1996; Nolte et al., 1997). En C. albicans, se ha demostrado que las mutaciones en ERG 2, 3, 5, 6 u 11 tienen este efecto (Arendrup y Patterson, 2017). Rhodes et al. examinaron 27 aislados de C. auris del Reino Unido en busca de SNPs en estos genes en cepas que mostraban una sensibilidad reducida a la AMB. Sin embargo, no se encontraron variantes que explicaran estas diferencias en la susceptibilidad al fármaco (Rhodes et al., 2018).
2.1.4. Mecanismos de resistencia a la flucitosina (5-Fluorocitosina)
La flucitosina es un análogo de los nucleósidos que inhibe la síntesis de ácidos nucleicos. Después de la entrada en la célula, la flucitosina tiene que ser activada para tener un efecto antifúngico. Esta activación requiere, entre otras, la proteína codificada por el gen FUR1 Waldorf y Polak (1983). En especies de Candida no auris se demostró que las mutaciones en FUR1 están relacionadas con la resistencia a la flucitosina (Vandeputte et al., 2012).
Rhodes et al. secuenciaron una cepa de C. auris resistente a la flucitosina y observaron una sustitución de aminoácidos F211I en el gen FUR1 (Rhodes et al., 2018). Esta mutación específica con sentido erróneo no tiene un equivalente conocido en otras especies de Candida, por lo que se requieren estudios adicionales para determinar si esta mutación es la causa de la resistencia a la flucitosina en la cepa de C. auris analizada (Rhodes et al., 2018). Sin embargo, este fármaco es menos empleado que otros antifúngicos. Por ello, se han realizado menos estudios para entender mejor la resistencia de C. auris a este compuesto.
Para resumir, algunos mecanismos de resistencia descubiertos previamente en especies de Candida como C. glabrata y C. albicans, también fueron mostrados por C. auris. Se enumeran en la última columna de la Tabla 1. Sin embargo, la resistencia antifúngica de C. auris sólo se explica parcialmente por estos mecanismos de resistencia previamente descritos. Con el fin de diseñar eventualmente nuevos agentes terapéuticos y, en general, de mejorar la atención a los pacientes, se necesitan más estudios para comprender mejor los mecanismos subyacentes que conducen a los rangos de CIM excepcionalmente altos que presentan algunos C. auris (véase la figura 1B).
2.2. Biofilm: Una forma de vida resistente a los antifúngicos
Se demostró que las CIM para varios antifúngicos son más altas en las células sésiles de C. auris (hasta 4, 20 y 60 veces para voriconazol, anfotericina B y micafungina, respectivamente) en comparación con las células planctónicas (Sherry et al., 2017). Del mismo modo, se demostró que las concentraciones mínimas de erradicación de biopelículas eran 512 veces superiores a las CIM para las equinocandinas y los azoles (Romera et al., 2019). Este fenómeno se demostró previamente para C. albicans (Hawser y Douglas, 1995). Los mecanismos moleculares responsables de estas elevadas CIM aún se desconocen en gran medida en C. auris, pero algunos estudios ya han proporcionado algunas ideas.
Se encontró que los genes que codifican la bomba de eflujo ABC están regulados al alza (de 2 a 4 veces) en las células sésiles en comparación con las células planctónicas (Kean et al., 2018a). Concordantemente, la actividad de las proteínas correspondientes se multiplicó por 2. Sin embargo, cuando se trató con inhibidores de la bomba de eflujo, la sensibilidad antifúngica de las células sésiles aumentó de 4 a 16 veces después de 12 h. Esto sugiere que las bombas de eflujo desempeñan un papel importante en la resistencia a los antifúngicos mostrada por este tipo de células (Kean et al., 2018a).
En C. albicans, se sabe que la matriz de exopolímeros se une de forma no específica a todas las clases de antifúngicos y los secuestra fuera de las células (Nett et al., 2010). Como Candida spp. comparten un perfil de polisacáridos central, se sugirió que este mecanismo podría estar conservado también en C. auris (Kean et al., 2018a). Esto se confirmó posteriormente en ensayos in vivo e in vitro (Domínguez et al., 2019).
Persistencia en el medio hospitalario
La capacidad de C. auris para colonizar y persistir en superficies es excepcional: es capaz de persistir más tiempo en superficies húmedas en comparación con C. albicans (Piedrahita et al., 2017). También tiene una actividad metabólica prolongada en las superficies, similar a la de C. parapsilosis, un conocido colonizador de la piel y el plástico (Piedrahita et al., 2017; Welsh et al., 2017).
La mayor supervivencia de C. auris en las superficies fue investigada por dos estudios. El primero afirmaba que podría estar relacionado con una mayor resistencia al estrés ambiental por la temperatura y otros factores de estrés (Kean et al., 2018b). El segundo plantea la hipótesis de que podría deberse a la formación de biofilm. Sin embargo, esto está por demostrar ya que, hasta ahora, no se recuperó ningún aislado formador de biofilm en las superficies ambientales (Ku et al., 2018). Cabe señalar que estas dos posibilidades no son mutuamente excluyentes, y que la formación de biofilm podría incluso ser la causa del aumento de la resistencia al estrés (Ku et al., 2018).
La persistencia de C. auris en entornos nosocomiales se ve aumentada por la resistencia de este patógeno a los procedimientos de desinfección. Se probó la eficacia del NaOCl y del ácido peracético en superficies de acero inoxidable, de polímero (cubreobjetos de poliéster) y de celulosa. Ambos desinfectantes mostraron una eficacia significativa para matar las células de C. auris en todas las superficies. Sin embargo, algunas células viables permanecieron después de aplicar NaOCl en superficies no porosas (acero inoxidable y cubreobjetos de poliéster). De hecho, se necesitaron concentraciones más altas de este desinfectante junto con tiempos de exposición más prolongados para reducir el rebrote, pero incluso así, no fueron capaces de erradicar completamente el patógeno. Por ejemplo, una concentración de 10.000 partes por millón (ppm) de NaOCl unida a una exposición de 5 minutos en acero inoxidable, fue suficiente para observar una reducción significativa de las colonias. Sin embargo, se observó un rebrote sustancial después de la reinoculación en un medio rico (Kean et al., 2018b).
Para el ácido peracético, que es un desinfectante de alto nivel, este rebrote dependía de la superficie, ya que no se observó ningún rebrote para el polímero a diferencia del acero inoxidable. Realizaron los mismos experimentos con C. glabrata y C. albicans y obtuvieron resultados similares (Kean et al., 2018b).
Esto demuestra que los procedimientos de desinfección de alto nivel pueden ser insuficientes para una limpieza completa de la superficie y que las directrices preestablecidas para la desinfección de superficies tienen que adaptarse a estas especies de Candida (Kean et al., 2018b). A la hora de aplicar estos procedimientos de desinfección deben tenerse en cuenta factores como el tipo de superficies y los tiempos de exposición (Kean et al., 2018b; Ku et al., 2018) y no solo el coste y la facilidad de uso, como suele ocurrir (Ku et al., 2018).
A partir de los resultados de dos estudios (Cadnum et al., 2017; Rutala et al., 2019), los CDC recomiendan el uso de desinfectantes eficaces contra las esporas de Clostridioides difficile. Si no es posible, sugieren alternativas como peróxido de hidrógeno al 0,5-1,4% o compuestos de amonio cuaternario complementados con alcohol isopropílico y/o alcohol etílico2. Por último, la luz ultravioleta C también podría ser un candidato interesante para la desinfección de superficies. Se ha demostrado que es muy eficaz para eliminar las colonias de C. auris, siempre que el tiempo de exposición y la distancia sean suficientes. Sin embargo, sería necesario realizar más estudios antes de aplicar estos métodos en entornos hospitalarios (de Groot et al., 2019).
Conclusión
Las capacidades patógenas y virulentas de C. auris son profundamente preocupantes. Sobre todo porque los brotes se produjeron de forma independiente en diferentes regiones del mundo y que los aislados asociados mostraron una menor susceptibilidad a los fármacos antifúngicos más utilizados para el tratamiento de los pacientes (ver Figura 1).
Para explicar la menor susceptibilidad antifúngica en C. auris, los investigadores hicieron uso de los amplios conocimientos ya disponibles sobre otras especies de Candida, particularmente C. albicans. Como resultado, se destacaron algunos de los mecanismos de resistencia de C. auris (véase la Tabla 1). Más concretamente, se comprobó que dos mutaciones puntuales en ERG11 y la sobreexpresión del transportador ABC Cdr1 disminuyen la sensibilidad al fluconazol. Además, se demostró que una sustitución de aminoácidos en FKS1 reduce la sensibilidad de C. auris a las equinocandinas, que normalmente es el fármaco de elección para el tratamiento contra este patógeno (Bidaud et al., 2018). Por último, se identificó una mutación en FUR1 en una cepa de C. auris resistente a la flucitosina, pero queda por demostrar que es la causa de esta sensibilidad reducida. Se cree que el biofilm es un mecanismo de resistencia en sí mismo, ya que se demostró que disminuye la susceptibilidad al fármaco. Sin embargo, todavía es necesario seguir investigando para describir toda la variedad de procesos que tienen lugar en esta forma de crecimiento. Es importante señalar que el biofilm es una estructura compleja que sigue siendo poco conocida y difícil de investigar, incluso en especies bien conocidas como C. albicans.
Las propiedades de resistencia de C. auris van unidas a una propensión a causar infecciones nosocomiales. Su capacidad para adherirse a una variedad de superficies nosocomiales y sobrevivir a la desinfección son factores críticos de propagación. La presencia de múltiples tipos de superficies supone un reto para las instituciones clínicas, ya que los procedimientos estándar de desinfección son ineficaces y no están adaptados a cada tipo de ellas. Este problema, también se deriva de la insuficiente investigación sobre este tema. Además, una mejor comprensión de los mecanismos que subyacen a la resistencia de C. auris a los desinfectantes ayudaría a mejorar los protocolos de higiene y a evitar futuros brotes nosocomiales.
Contribuciones de los autores
FC, AG y LJ participaron por igual en la redacción de esta revisión. AC supervisó y participó en la redacción de esta revisión.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Agradecimientos
Deseamos agradecer a Clara Heiman como hablante nativa de inglés por haber revisado nuestro manuscrito.
Notas a pie de página
1. ^CDC (2019). Pruebas de susceptibilidad a los antifúngicos e interpretación. Disponible en línea en: https://www.cdc.gov/fungal/candida-auris/c-auris-antifungal.html (consultado el 29 de abril de 2019).
2. ^CDC (2018). Prevención y control de infecciones por Candida auris. Disponible en línea en: https://www.cdc.gov/fungal/candida-auris/c-auris-infection-control.html (consultado el 12 de noviembre de 2019).
Abdalhamid, B., Almaghrabi, R., Althawadi, S., y Omrani, A. (2018). Primer informe de infecciones por Candida auris en Arabia Saudí. J. Infect. Public Health 11, 598-599. doi: 10.1016/j.jiph.2018.05.010
PubMed Abstract | CrossRef Full Text | Google Scholar
Adam, R. D., Revathi, G., Okinda, N., Fontaine, M., Shah, J., Kagotho, E., et al. (2019). Análisis de la fungemia por Candida auris en un único centro de Kenia. Int. J. Infect. Dis. 85, 182-187. doi: 10.1016/j.ijid.2019.06.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Adams, E., Quinn, M., Tsay, S., Poirot, E., Chaturvedi, S., Southwick, K., et al. (2018). Candida auris en instalaciones sanitarias, Nueva York, Estados Unidos, 2013-2017. Emerg. Infect. Dis. 24, 1816-1824. doi: 10.3201/eid2410.180649
CrossRef Full Text | Google Scholar
Arendrup, M. C., y Patterson, T. F. (2017). Candida multirresistente: epidemiología, mecanismos moleculares y tratamiento. J. Infect. Dis. 216, S445-S451. doi: 10.1093/infdis/jix131
PubMed Abstract | CrossRef Full Text | Google Scholar
Barantsevich, N. E., Orlova, O. E., Shlyakhto, E. V., Johnson, E. M., Woodford, N., Lass-Floerl, C., et al. (2019). Emergencia de Candida auris en Rusia. J. Hosp. Infect. 102, 445-448. doi: 10.1016/j.jhin.2019.02.021
PubMed Abstract | CrossRef Full Text | Google Scholar
Belkin, A., Gazit, Z., Keller, N., Ben-Ami, R., Wieder-Finesod, A., Novikov, A., et al. (2018). Infección por Candida auris que conduce a la transmisión nosocomial, Israel, 2017. Emerg. Infect. Dis. 24, 801-804. doi: 10.3201/eid2404.171715
PubMed Abstract | CrossRef Full Text | Google Scholar
Ben-Ami, R., Berman, J., Novikov, A., Bash, E., Shachor-Meyouhas, Y., Zakin, S., et al. (2017). Candida haemulonii y C. auris multirresistentes, Tel Aviv, Israel. Emerg. Infect. Dis. 23, 195-203. doi: 10.3201/eid2302.161486
PubMed Abstract | CrossRef Full Text | Google Scholar
Bidaud, A. L., Chowdhary, A., y Dannaoui, E. (2018). Candida auris: una levadura emergente resistente a los medicamentos – Una mini-revisión. J. Mycol. Méd. 28, 568-573. doi: 10.1016/j.mycmed.2018.06.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Biswal, M., Rudramurthy, S. M., Jain, N., Shamanth, A. S., Sharma, D., Jain, K., et al. (2017). Control de un posible brote de infección por Candida auris: lecciones aprendidas de múltiples intervenciones. J. Hosp. Infect. 97, 363-370. doi: 10.1016/j.jhin.2017.09.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Borman, A. M., Szekely, A., y Johnson, E. M. (2016). Patogenicidad comparativa de los aislados del Reino Unido del patógeno emergente Candida auris y otras especies clave de cándida patógena. mSphere 1:e00189-16. doi: 10.1128/mSphere.00189-16
CrossRef Full Text | Google Scholar
Cadnum, J. L., Shaikh, A. A., Piedrahita, C. T., Sankar, T., Jencson, A. L., Larkin, E. L., et al. (2017). Eficacia de los desinfectantes contra Candida auris y otras especies de cándida. Infect. Control Hosp. Epidemiol. 38, 1240-1243. doi: 10.1017/ice.2017.162
PubMed Abstract | CrossRef Full Text | Google Scholar
Chatterjee, S., Alampalli, S. V., Nageshan, R. K., Chettiar, S. T., Joshi, S., y Tatu, U. S. (2015). Borrador del genoma de un patógeno comúnmente mal diagnosticado resistente a los medicamentos Candida auris. BMC Genomics 16:686. doi: 10.1186/s12864-015-1863-z
PubMed Abstract | CrossRef Full Text | Google Scholar
Chow, N. A., Gade, L., Tsay, S. V., Forsberg, K., Greenko, J. A., Southwick, K. L., et al. (2018). Múltiples introducciones y posterior transmisión de Candida auris multirresistente en los Estados Unidos: un estudio epidemiológico molecular. Lancet Infect. Dis. 18, 1377-1384. doi: 10.1016/S1473-3099(18)30597-8
PubMed Abstract | CrossRef Full Text | Google Scholar
Chowdhary, A., Prakash, A., Sharma, C., Kordalewska, M., Kumar, A., Sarma, S., et al. (2018). Un estudio multicéntrico de los patrones de susceptibilidad antifúngica entre 350 aislados de Candida auris (2009-17) en la India: papel de los genes ERG11 y FKS1 en la resistencia a los azoles y las equinocandinas. J. Antimicrob. Chemother. 73, 891-899. doi: 10.1093/jac/dkx480
PubMed Abstract | CrossRef Full Text | Google Scholar
de Groot, T., Chowdhary, A., Meis, J. F., y Voss, A. (2019). Matanza de Candida auris por UV-C: importancia del tiempo y la distancia de exposición. Mycoses 62, 408-412. doi: 10.1111/myc.12903
PubMed Abstract | CrossRef Full Text | Google Scholar
Desoubeaux, G., Bailly, E., Guillaume, C., De Kyvon, M.-A., Tellier, A.-C., Morange, V., et al. (2018). Candida auris en los laboratorios de micología contemporáneos: algunos trucos prácticos para identificarla de forma fiable según una experiencia francesa reciente. J. Mycol. Med. 28, 407-410. doi: 10.1016/j.mycmed.2018.02.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Domínguez, E. G., Zarnowski, R., Choy, H. L., Zhao, M., Sánchez, H., Nett, J. E., et al. (2019). Papel conservado de los polisacáridos de la matriz de la biopelícula en la resistencia a los medicamentos de Candida auris. mSphere 4:e00680-18. doi: 10.1128/mSphereDirect.00680-18
Abstracto de PubMed | Texto Completo de Google | Google Scholar
Escandón, P., Chow, N. A., Caceres, D. H., Gade, L., Berkow, E. L., Armstrong, P., et al. (2019). Epidemiología molecular de Candida auris en colombia revela una colonización altamente relacionada, a nivel nacional, con patrones regionales en la resistencia a la anfotericina B. Clin. Infect. Dis. 68, 15-21. doi: 10.1093/cid/ciy411
CrossRef Full Text | Google Scholar
Eyre, D. W., Sheppard, A. E., Madder, H., Moir, I., Moroney, R., Quan, T. P., et al. (2018). Un brote de Candida auris y su control en un entorno de cuidados intensivos. N. Engl. J. Med. 379, 1322-1331. doi: 10.1056/NEJMoa1714373
PubMed Abstract | CrossRef Full Text | Google Scholar
Fakhim, H., Vaezi, A., Dannaoui, E., Chowdhary, A., Nasiry, D., Faeli, L., et al. (2018). Virulencia comparativa de Candida auris con Candida haemulonii, Candida glabrata y Candida albicans en un modelo murino. Mycoses 61, 377-382. doi: 10.1111/myc.12754
PubMed Abstract | CrossRef Full Text | Google Scholar
Hawser, S. P., y Douglas, L. J. (1995). Resistencia de las biopelículas de Candida albicans a los agentes antifúngicos in vitro. Antimicrob. Agents Chemother. 39, 2128-2131. doi: 10.1128/AAC.39.9.2128
PubMed Abstract | CrossRef Full Text | Google Scholar
Haynes, M. P., Chong, P. L.-G., Buckley, H. R., y Pieringer, R. A. (1996). Estudios de fluorescencia sobre la acción molecular de la anfotericina B en células fúngicas susceptibles y resistentes. Biochemistry 35, 7983-7992. doi: 10.1021/bi952910c
PubMed Abstract | CrossRef Full Text | Google Scholar
Healey, K. R., Kordalewska, M., Ortigosa, C. J., Singh, A., Berrío, I., Chowdhary, A., et al. (2018). Las mutaciones limitadas de ERG11 identificadas en aislados de Candida auris contribuyen directamente a una menor susceptibilidad a los azoles. Antimicrob. Agents Chemother. 62, e01427-18. doi: 10.1128/AAC.01427-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Jackson, B. R., Chow, N., Forsberg, K., Litvintseva, A. P., Lockhart, S. R., Welsh, R., et al. (2019). Sobre los orígenes de una especie: ¿qué podría explicar el surgimiento de Candida auris? J. Fungi 5:E58. doi: 10.3390/jof5030058
PubMed Abstract | CrossRef Full Text | Google Scholar
Kean, R., Delaney, C., Sherry, L., Borman, A., Johnson, E. M., Richardson, M. D., et al. (2018a). El ensamblaje y el perfil del transcriptoma de Candida auris revelan nuevos conocimientos sobre la resistencia mediada por la biopelícula. mSphere 3:e00334-18. doi: 10.1128/mSphere.00334-18
Abstracto de PubMed | Texto completo de la encuesta de Google
Kean, R., Sherry, L., Townsend, E., McKloud, E., Short, B., Akinbobola, A., et al. (2018b). Desafíos de desinfección de superficies para Candida auris: un estudio in vitro. J. Hosp. Infect. 98, 433-436. doi: 10.1016/j.jhin.2017.11.015
PubMed Abstract | CrossRef Full Text | Google Scholar
Kenters, N., Kiernan, M., Chowdhary, A., Denning, D. W., Pemán, J., Saris, K., et al. (2019). Control de Candida auris en instituciones sanitarias. Resultado de una reunión de expertos del ISAC. Int. J. Antimicrob. Agents 54, 400-406. doi: 10.1016/j.ijantimicag.2019.08.013
CrossRef Full Text | Google Scholar
Kordalewska, M., Lee, A., Park, S., Berrio, I., Chowdhary, A., Zhao, Y., et al. (2018). Entendiendo la resistencia a las equinocandinas en el patógeno emergente Candida auris. Antimicrob. Agents Chemother. 62:e00238-18. doi: 10.1128/AAC.00238-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Krishnasamy, L., Krishnakumar, S., Kumaramanickavel, G., y Saikumar, C. (2018). Mecanismos moleculares de la resistencia a los medicamentos antifúngicos en especies de Candida. J. Clin. Diagn. Res. 12, DE01-DE06. doi: 10.7860/JCDR/2018/36218.11961
CrossRef Full Text | Google Scholar
Ku, T. S. N., Walraven, C. J., y Lee, S. A. (2018). Candida auris: desinfectantes e implicaciones para el control de infecciones. Front. Microbiol. 9:726. doi: 10.3389/fmicb.2018.00726
CrossRef Full Text | Google Scholar
Leach, L., Russell, A., Zhu, Y., Chaturvedi, S., y Chaturvedi, V. (2019). Un ensayo de PCR en tiempo real de Candida auris rápido y automatizado para el análisis de alto rendimiento de muestras de vigilancia con el sistema abierto BD MAX™. J. Clin. Microbiol. 57:e00630-19. doi: 10.1128/JCM.00630-19
CrossRef Full Text | Google Scholar
Lesho, E. P., Bronstein, M. Z., McGann, P., Stam, J., Kwak, Y., Maybank, R., et al. (2018). Importación, mitigación y epidemiología genómica de Candida auris en un gran hospital universitario. Infect. Control Hosp. Epidemiol. 39, 53-57. doi: 10.1017/ice.2017.231
PubMed Abstract | CrossRef Full Text | Google Scholar
Lockhart, S. R., Etienne, K. A., Vallabhaneni, S., Farooqi, J., Chowdhary, A., Govender, N. P., et al. (2017). Emergencia simultánea de Candida auris multirresistente en 3 continentes confirmada por la secuenciación del genoma completo y los análisis epidemiológicos. Clin. Infect. Dis. 64, 134-140. doi: 10.1093/cid/ciw691
PubMed Abstract | CrossRef Full Text | Google Scholar
Lopez-Ribot, J. L., McAtee, R. K., Lee, L. N., Kirkpatrick, W. R., White, T. C., Sanglard, D., et al. (1998). Distinct patterns of gene expression associated with development of fluconazole resistance in serial Candida albicans isolates from human immunodeficiency virus-infected patients with oropharyngeal candidiasis. Antimicrob. Agents Chemother. 42, 2932-2937. doi: 10.1128/AAC.42.11.2932
PubMed Abstract | CrossRef Full Text | Google Scholar
Martins, I. M., Cortés, J. C. G., Muñoz, J., Moreno, M. B., Ramos, M., Clemente-Ramos, J. A., et al. (2011). Actividades diferenciales de tres familias de inhibidores específicos de la beta(1,3)glucano sintasa en cepas de levadura de fisión de tipo salvaje y resistentes. J. Biol. Chem. 286, 3484-3496. doi: 10.1074/jbc.M110.174300
PubMed Abstract | CrossRef Full Text | Google Scholar
Mayor, S. (2018). El brote de Candida auris estuvo vinculado a las sondas de temperatura axilar reutilizables, según un estudio. BMJ 363:k4133. doi: 10.1136/bmj.k4133
PubMed Abstract | CrossRef Full Text | Google Scholar
Morschhäuser, J., Barker, K. S., Liu, T. T., Bla-Warmuth, J., Homayouni, R., y Rogers, P. D. (2007). El factor de transcripción Mrr1p controla la expresión de la bomba de eflujo MDR1 y media la resistencia a múltiples fármacos en Candida albicans. PLoS Pathog. 3:e164. doi: 10.1371/journal.ppat.0030164
PubMed Abstract | CrossRef Full Text | Google Scholar
Nett, J. E., Crawford, K., Marchillo, K., y Andes, D. R. (2010). Role of Fks1p and matrix glucan in Candida albicans biofilm resistance to an echinocandin, pyrimidine, and polyene. Antimicrob. Agents Chemother. 54, 3505-3508. doi: 10.1128/AAC.00227-10
PubMed Abstract | CrossRef Full Text | Google Scholar
Nolte, F. S., Parkinson, T., Falconer, D. J., Dix, S., Williams, J., Gilmore, C., et al. (1997). Isolation and characterization of fluconazole- and amphotericin B-resistant Candida albicans from blood of two patients with leukemia. Antimicrob. Agents Chemother. 41, 196-199. doi: 10.1128/AAC.41.1.196
PubMed Abstract | CrossRef Full Text | Google Scholar
O’Connor, C., Bicanic, T., Dave, J., Evans, T. J., Moxey, P., Adamu, U., et al. (2019). Brote de Candida auris en una sala vascular: la llegada inesperada de un patógeno anticipado. J. Hosp. Infect. 103, 106-108. doi: 10.1016/j.jhin.2019.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Osei Sekyere, J. (2018). Candida auris: una revisión sistemática y un metaanálisis de las actualizaciones actuales sobre un patógeno emergente multirresistente. MicrobiologyOpen 7:e00578. doi: 10.1002/mbo3.578
PubMed Abstract | CrossRef Full Text | Google Scholar
Park, J. Y., Bradley, N., Brooks, S., Burney, S., y Wassner, C. (2019). Manejo de pacientes con fungemia por Candida auris en el hospital comunitario, Brooklyn, Nueva York, Estados Unidos, 2016-20181. Emerg. Infect. Dis. 25, 601-602. doi: 10.3201/eid2503.180927
PubMed Abstract | CrossRef Full Text | Google Scholar
Park, S., Kelly, R., Kahn, J. N., Robles, J., Hsu, M.-J., Register, E., et al. (2005). Specific substitutions in the echinocandin target Fks1p account for reduced susceptibility of rare laboratory and clinical Candida sp. isolates. Antimicrob. Agents Chemother. 49, 3264-3273. doi: 10.1128/AAC.49.8.3264-3273.2005
CrossRef Full Text | Google Scholar
Parra-Giraldo, C. M., Valderrama, S. L., Cortes-Fraile, G., Garzón, J. R., Ariza, B. E., Morio, F., et al. (2018). Primer reporte de casos esporádicos de Candida auris en Colombia. Int. J. Infect. Dis. 69, 63-67. doi: 10.1016/j.ijid.2018.01.034
PubMed Abstract | CrossRef Full Text | Google Scholar
Pemán, J., Cantón, E., y Espinel-Ingroff, A. (2009). Mecanismos de resistencia a fármacos antifúngicos. Expert Rev. Anti Infect. Ther. 7, 453-460. doi: 10.1586/eri.09.18
PubMed Abstract | CrossRef Full Text | Google Scholar
Piedrahita, C. T., Cadnum, J. L., Jencson, A. L., Shaikh, A. A., Ghannoum, M. A., and Donskey, C. J. (2017). Las superficies ambientales en los centros sanitarios son una fuente potencial de transmisión de Candida auris y otras especies de cándida. Infect. Control Hosp. Epidemiol. 38, 1107-1109. doi: 10.1017/ice.2017.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Rhodes, J., Abdolrasouli, A., Farrer, R. A., Cuomo, C. A., Aanensen, D. M., Armstrong-James, D., et al. (2018). Epidemiología genómica del brote del Reino Unido del patógeno fúngico humano emergente Candida auris. Emerg. Microbes Infect. 7:43. doi: 10.1101/201343
CrossRef Full Text | Google Scholar
Romera, D., Aguilera-Correa, J. J., Gadea, I., Viñuela-Sandoval, L., García-Rodríguez, J., y Esteban, J. (2019). Candida auris: comparación entre la susceptibilidad planctónica y en biofilm a los antifúngicos. J. Med. Microbiol. 68, 1353-1358. doi: 10.1099/jmm.0.001036
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A., Martínez, H., Moret, A. M., Calabuig, E., Tasias, M., Alastruey-Izquierdo, A., et al. (2019a). Detección y tratamiento de Candida auris en situación de brote: factores de riesgo para desarrollar colonización y candidemia por esta nueva especie en pacientes críticos. Expert Rev. Anti Infect. Ther. 17, 295-305. doi: 10.1080/14787210.2019.1592675
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A., Moret, A. M., Tasias-Pitarch, M., Aleixandre-López, A. I., Martínez-Morel, H., Calabuig, E., et al. (2018). Un brote debido a Candida auris con colonización prolongada y candidemia en un hospital europeo de atención terciaria. Micosis 61, 498-505. doi: 10.1111/myc.12781
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A. C., Cantón, E., Fernández-Rivero, M. E., Ramírez, P., y Pemán, J. (2019b). Brote de Candida auris en España: comparación de la actividad antifúngica por tres métodos con los datos publicados. Int. J. Antimicrob. Agents 53, 541-546. doi: 10.1016/j.ijantimicag.2019.02.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Rutala, W. A., Kanamori, H., Gergen, M. F., Sickbert-Bennett, E. E., y Weber, D. J. (2019). Susceptibilidad de Candida auris y Candida albicans a 21 germicidas utilizados en instalaciones sanitarias. Infect. Control Hosp. Epidemiol. 40, 380-382. doi: 10.1017/ice.2019.1
PubMed Abstract | CrossRef Full Text | Google Scholar
Rybak, J. M., Doorley, L. A., Nishimoto, A. T., Barker, K. S., Palmer, G. E., and Rogers, P. D. (2019). Abrogación de la resistencia al triazol tras la deleción de CDR1 en un aislado clínico de Candida auris. Antimicrob. Agents Chemother. 63, e00057-19. doi: 10.1128/AAC.00057-19
PubMed Abstract | CrossRef Full Text | Google Scholar
Sana, F., Hussain, W., Zaman, G., Satti, L., Khurshid, U., y Khadim, M. T. (2019). Informe del brote de Candida auris en Pakistán: una historia de éxito en el control de infecciones en las UCI de un hospital de atención terciaria. J. Hosp. Infect. 103, 108-110. doi: 10.1016/j.jhin.2019.06.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Sanglard, D., Coste, A., y Ferrari, S. (2009). Antifungal drug resistance mechanisms in fungal pathogens from the perspective of transcriptional gene regulation. FEMS Yeast Res. 9, 1029-1050. doi: 10.1111/j.1567-1364.2009.00578.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Sanglard, D., Ischer, F., Koymans, L., and Bille, J. (1998). Amino acid substitutions in the cytochrome P-450 lanosterol 14-demethylase (CYP51a1) from azole-resistant Candida albicans clinical isolates contribute to resistance to azole antifungal agents. Antimicrob. Agents Chemother. 42, 241-253.
Google Scholar
Sanglard, D., Kuchler, K., Ischer, F., Pagani, J. L., Monod, M., y Bille, J. (1995). Mechanisms of resistance to azole antifungal agents in Candida albicans isolates from AIDS patients involve specific multidrug transporters. Antimicrob. Agents Chemother. 39, 2378-2386. doi: 10.1128/AAC.39.11.2378
PubMed Abstract | CrossRef Full Text | Google Scholar
Sayeed, M. A., Farooqi, J., Jabeen, K., Awan, S., y Mahmood, S. F. (2019). Espectro clínico y factores que impactan el resultado de Candida auris: un estudio de un solo centro de Pakistán. BMC Infect. Dis. 19:384. doi: 10.1186/s12879-019-3999-y
PubMed Abstract | CrossRef Full Text | Google Scholar
Schelenz, S., Hagen, F., Rhodes, J. L., Abdolrasouli, A., Chowdhary, A., Hall, A., et al. (2016). Primer brote hospitalario de Candida auris, emergente a nivel mundial, en un hospital europeo. Antimicrob. Resist. Infect. Control 5:35. doi: 10.1186/s13756-016-0132-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Schuetzer-Muehlbauer, M., Willinger, B., Egner, R., Ecker, G., y Kuchler, K. (2003). Reversión de la resistencia antifúngica mediada por bombas de eflujo ABC de Candida albicans expresadas funcionalmente en la levadura. Int. J. Antimicrob. Agents 22, 291-300. doi: 10.1016/S0924-8579(03)00213-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Sexton, D. J., Kordalewska, M., Bentz, M. L., Welsh, R. M., Perlin, D. S., y Litvintseva, A. P. (2018). Detección directa del patógeno fúngico emergente Candida auris en hisopos cutáneos clínicos mediante un ensayo de PCR cuantitativo basado en SYBR green. J. Clin. Microbiol. 56:e01337-18. doi: 10.1128/JCM.01337-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sharma, C., Kumar, N., Pandey, R., Meis, J. F., y Chowdhary, A. (2016). Whole genome sequencing of emerging multidrug resistant Candida auris isolates in India demonstrates low genetic variation. New Microbes New Infect. 13, 77-82. doi: 10.1016/j.nmni.2016.07.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Sherry, L., Ramage, G., Kean, R., Borman, A., Johnson, E. M., Richardson, M. D., et al. (2017). Capacidad de formación de biopelículas de Candida auris altamente virulenta y multirresistente. Emerg. Infect. Dis. 23, 328-331. doi: 10.3201/eid2302.161320
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandeputte, P., Ferrari, S., and Coste, A. T. (2012). Resistencia a los antifúngicos y nuevas estrategias para controlar las infecciones fúngicas. Int. J. Microbiol. 2012:713687. doi: 10.1155/2012/713687
PubMed Abstract | CrossRef Full Text | Google Scholar
Waldorf, A. R., y Polak, A. (1983). Mechanisms of action of 5-fluorocytosine. Antimicrob. Agents Chemother. 23, 79-85. doi: 10.1128/AAC.23.1.79
PubMed Abstract | CrossRef Full Text | Google Scholar
Wang, X., Bing, J., Zheng, Q., Zhang, F., Liu, J., Yue, H., et al. (2018). El primer aislado de Candida auris en China: aspectos clínicos y biológicos. Emerging Microbes & Infections 7, 1-9. doi: 10.1038/s41426-018-0095-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Welsh, R. M., Bentz, M. L., Shams, A., Houston, H., Lyons, A., Rose, L. J., et al. (2017). Supervivencia, persistencia y aislamiento de la levadura patógena emergente multirresistente Candida auris en una superficie sanitaria de plástico. J. Clin. Microbiol. 55, 2996-3005. doi: 10.1128/JCM.00921-17
PubMed Abstract | CrossRef Full Text | Google Scholar
Leave a Reply