Frontiers in Microbiology
Introduction
In den letzten zehn Jahren hat eine nosokomiale Candida-Spezies schwere Fälle von Candidämie und andere Arten von Pilzinfektionen verursacht. Ausbrüche des verantwortlichen Erregers, nämlich Candida auris, wurden auf der ganzen Welt beobachtet (siehe Abbildung 1A) (Lockhart et al., 2017).
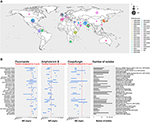
Abbildung 1. (A) Karte mit der Anzahl der Isolate pro Ausbruch seit 1996. Das erste Ausbruchsjahr ist braun und das letzte Ausbruchsjahr rosa eingefärbt. Die Größe der Kreise ist proportional zur Anzahl der für jeden Ausbruch gemeldeten Isolate. Die Erhebung der Ausbrüche wurde anhand von Daten aus drei Übersichtsarbeiten (Osei Sekyere, 2018; Jackson et al., 2019; Kenters et al., 2019) durchgeführt und durch eine pubmed-Suche mit den Stichworten „Candida“ AND „auris“ AND „outbreak“ ergänzt. Die Ergebnisse waren zeitlich begrenzt und lagen zwischen dem 21.07.2017, der letzten Studie in der systematischen Übersichtsarbeit von Osei Sekyere, und dem 31.08.2019, als die Literatursuche durchgeführt wurde. Studien, die nicht in der Übersichtsarbeit von Osei Sekyere enthalten sind: (Biswal et al., 2017; Abdalhamid et al., 2018; Adams et al., 2018; Belkin et al., 2018; Chow et al., 2018; Desoubeaux et al., 2018; Lesho et al., 2018; Mayor, 2018; Parra-Giraldo et al., 2018; Ruiz-Gaitán et al, 2018, 2019a,b; Sexton et al., 2018; Adam et al., 2019; Barantsevich et al., 2019; Escandón et al., 2019; Leach et al., 2019; O’Connor et al., 2019; Park et al., 2019; Sana et al., 2019; Sayeed et al., 2019). (B) MHK-Bereiche für drei Antimykotika, die bei C. auris-Isolaten aus verschiedenen Ausbrüchen auf der ganzen Welt gemessen wurden. Die rote gepunktete Linie stellt den von der CDC vorgeschlagenen vorläufigen Breakpoint dar. IPSV ist ein Akronym für „Indien, Pakistan, Südafrika und Venezuela“, da Stämme aus diesen Ländern in derselben Studie analysiert wurden. Die Ausbruchsuntersuchung wurde identisch zu (A) durchgeführt. Es wurden nur Ausbrüche mit >1 Isolat aufgezeichnet. Von Osei Sekyere zitierte Studien sind mit und von Kenters et al. zitierte Studien mit gekennzeichnet.
Die Sequenzierung des gesamten Genoms ergab das Vorhandensein von vier spezifischen Kladen innerhalb der C. auris-Spezies: Ostasien, Südasien, Südafrika und Südamerika. Alle Kladen sind durch unterschiedliche Einzelnukleotid-Polymorphismen (SNPs) charakterisiert, was das unabhängige und weltweite Auftreten dieses Erregers unterstreicht (Lockhart et al., 2017).
Vergleichende Studien in verschiedenen Tiermodellen (Mäuse- und Galleria mellonella-Larven) zeigten stammabhängige Virulenzniveaus für C. auris. In den meisten Fällen erwiesen sich C. auris-Stämme als weniger virulent als C. albicans (Borman et al., 2016; Ben-Ami et al., 2017; Fakhim et al., 2018; Wang et al., 2018). Allerdings waren die gemeldeten Fälle von C. auris-Ausbrüchen mit einer hohen Sterblichkeitsrate verbunden, vor allem bei immungeschwächten Patienten. Andere Erkrankungen wie Diabetes, kardiovaskuläre und/oder pulmonale Erkrankungen, Sepsis oder eine vorherige Behandlung mit Antibiotika schienen ebenfalls wichtige Risikofaktoren zu sein (Osei Sekyere, 2018).
Außergewöhnlich für einen Hefepilz hat sich C. auris erfolgreich in nosokomialen Umgebungen verbreitet. In einem britischen Krankenhaus beispielsweise führte ein einziger mit C. auris kolonisierter Patient zur Identifizierung weiterer Fälle bei anderen hospitalisierten Patienten. Der Erreger wurde auch noch mehrere Monate nach seiner ersten Isolierung beim Gesundheitspersonal nachgewiesen, was auf eine effiziente Übertragung von Mensch zu Mensch schließen lässt. Darüber hinaus wurden Spuren von C. auris auf Bettseiten, Fensterbänken, Monitoren und anderen Geräten im gesamten Krankenhaus gefunden, was zeigt, dass C. auris im Gegensatz zu anderen Candida-Arten in der Lage ist, auf nosokomialen Oberflächen zu persistieren (Schelenz et al., 2016). Bei der Untersuchung eines Ausbruchs in einem anderen Krankenhaus im Vereinigten Königreich wurde berichtet, dass die Verwendung von wiederverwendbaren Geräten wie axillären Temperatursonden die Hauptursache für die Übertragung auf Patienten war (Eyre et al., 2018), was die Fähigkeit von C. auris, auf Oberflächen zu überleben, weiter veranschaulicht.
Die Fähigkeit, in nosokomialen Umgebungen zu überleben, könnte auf die Fähigkeit von C. auris zurückzuführen sein, Biofilme zu bilden; eine Wachstumsform, bei der Zellen in Mikrokolonien angeordnet sind, die durch eine Glucanmatrix geschützt sind. Konventionell werden Zellen, die von der Matrix umschlossen sind, als sessil bezeichnet, während Zellen außerhalb der Matrix als planktonisch bezeichnet werden. Es hat sich gezeigt, dass C. auris-Zellen, die durch diese Struktur geschützt sind, selbst mit hochgradigen Desinfektionsverfahren nur schwer zu beseitigen sind (Kean et al., 2018b).
Die Fähigkeit von C. auris, Biofilme zu bilden, ermöglicht nicht nur möglicherweise ein längeres Überleben auf inerten Oberflächen, sondern könnte auch ein wichtiger Faktor für seine Resistenz gegen systemische Antimykotika sein, die bei der Behandlung von Patienten eingesetzt werden. Die Tatsache, dass biofilmbildende Zellen aus Wunden und Katheterspitzen isoliert wurden, deutet in der Tat darauf hin, dass einige C. auris-Zellen in Biofilmform bei Patienten überleben könnten (Borman et al., 2016), und es wurde gezeigt, dass sessile Zellen eine verringerte Anfälligkeit für mehrere Antimykotika aufweisen. (Sherry et al., 2017). Schließlich hat sich der Biofilm auch als wichtiger Virulenzfaktor erwiesen, da biofilmbildende C. auris-Stämme mit einer erhöhten Morbidität und Mortalität in Verbindung gebracht werden (Borman et al, 2016).
Mit dieser Übersichtsarbeit wollen wir die von C. auris erworbenen molekularen Resistenzmechanismen beleuchten, die zu seinem außergewöhnlichen Überleben in nosokomialer Umgebung und bei hospitalisierten Patienten geführt haben.
C. auris Antimykotische Empfindlichkeit
Nach 10 Jahren der Aufzeichnung der minimalen Hemmkonzentration (MHK) für verfügbare Antimykotika hat die CDC kürzlich eine vorläufige Liste von Breakpoints veröffentlicht, um die antimykotische Empfindlichkeit von C. auris besser zu definieren1. Es zeigte sich deutlich, dass C. auris außergewöhnlich hohe MHKs für die vier wichtigsten Klassen von Antimykotika aufweist: Azole, Echinocandine, Polyene (Abbildung 1B) und Nukleosidanaloga. In diesem Kapitel werden zwei Faktoren eingehend untersucht, die nachweislich eine Rolle bei dieser verminderten Anfälligkeit für antimykotische Behandlungen spielen: molekulare Resistenzmechanismen und Biofilm-assoziierte Resistenzmechanismen.
2.1. Molekulare Resistenzmechanismen
Wir definieren hier molekulare Resistenz als die Art und Weise, wie einzelne Zellen ihre Empfindlichkeit gegenüber Antimykotika aufgrund von genetischen Veränderungen, die zu einer Veränderung des Wirkstoffziels, einer Überexpression von Effluxpumpen oder einer Veränderung des Stoffwechsels führen, verringern können (siehe für eine Übersicht: Pemán et al., 2009; Sanglard et al., 2009; Krishnasamy et al., 2018). In diesem Abschnitt werden für jede Klasse von Antimykotika die wichtigsten molekularen Resistenzmechanismen zusammengestellt, die bisher bei C. auris gefunden wurden. Zusätzlich fasst Tabelle 1 für jeden Wirkstoff die Wirkungsweise, die damit verbundenen Resistenzmechanismen, die bei Candida non auris beschrieben wurden, und die bisher bei C. auris entdeckten Resistenzmechanismen zusammen.

Tabelle 1. Pharmakodynamik der vier wichtigsten Antimykotika-Klassen und damit verbundene Resistenzmechanismen bei Candida spp. im Allgemeinen und C. auris im Besonderen.
2.1.1. Mechanismen der Resistenz gegen Azole
2.1.1.1. Überexpression von MFS- und ABC-Effluxpumpen
Effluxpumpen sind Proteine, die Komponenten durch die Zellmembran transportieren. Einige von ihnen können Medikamente aus der Zelle pumpen und so ihre Konzentration und Wirkung in der Zelle verringern. Es gibt zwei große Familien von Effluxpumpen, die an der Resistenz gegen Pilze beteiligt sind: ATP Binding Cassette (ABC) und Major Facilitator Superfamily (MFS) Transporter. Eine verstärkte Überexpression von Effluxpumpen ist einer der wichtigsten Resistenzmechanismen gegen Azole in pathogenen Candida-Arten (Schuetzer-Muehlbauer et al., 2003; Morschhäuser et al., 2007).
Indische Stämme von C. auris, die in zwei verschiedenen Studien sequenziert wurden, weisen zahlreiche Gene auf, die ortholog zu ABC- und MFS-Transportern von C. albicans sind (Chatterjee et al., 2015; Sharma et al., 2016). Interessanterweise zeigten C. auris-Stämme, die in Israel isoliert wurden, eine intrinsisch hohe ABC-Transporteraktivität im Vergleich zu C. glabrata und C. haemulonii, indem der Efflux eines fluoreszierenden Substrats (Rhodamin 6G) gemessen wurde (Ben-Ami et al., 2017).
In C. albicans ist CDR1 ein Gen, das für eine ABC-Effluxpumpe kodiert, die für ihre Rolle bei der Azolresistenz bekannt ist (Sanglard et al., 1995). Ein zu CDR1 homologes Gen wurde in C. auris gefunden. (Rybak et al., 2019). Die gleiche Studie zeigte auch, dass die Deletion dieses Gens die Anfälligkeit resistenter Stämme um das 64- bis 128-fache erhöhen kann (Rybak et al., 2019).
2.1.1.2. Punktmutationen in ERG11
Ergosterol ist ein wichtiger Membranbestandteil in Pilzen. In Candida spp. wird seine Biosynthese unter anderem durch das Enzym Lanosterol 14-alpha-Demethylase (LD) vermittelt, das durch das Gen ERG11 kodiert wird und Lanosterol in Ergosterol umwandelt. Die LD ist das primäre Ziel von Azolen, Antimykotika, die die Funktion des Enzyms hemmen und die Ergosterol-Biosynthese effektiv abschalten, wodurch die Membranintegrität beeinträchtigt wird (Sanglard et al., 1998).
Punktmutationen in ERG11 reduzieren nachweislich die Azolempfindlichkeit von Candida spp, insbesondere in drei „Hot-Spot“-Regionen, die sich zwischen den Aminosäuren 105-165, 266-287 und 405-488 befinden (Vandeputte et al., 2012).
Interessanterweise wurden nach dem Abrufen der Aminosäuresequenzen von 44 C. auris-Isolaten aus Indien 15 Missense-Mutationen gefunden, wenn sie mit der C. albicans-Wildtyp-ERG11-Sequenz abgeglichen wurden (Chowdhary et al., 2018). Fünf dieser Mutationen wurden bereits mit Azolresistenz bei C. albicans in Verbindung gebracht. Von diesen Varianten wurden zwei in jedem in dieser Studie getesteten resistenten Stamm gefunden, nämlich Y132F oder K143R (Chowdhary et al., 2018). Es kann auch festgestellt werden, dass sich diese Mutationen im ersten „Hot Spot“ befinden, der zuvor diskutiert wurde.
Ein Screening nach Varianten im ERG11-Gen von C. auris-Stämmen aus Kolumbien identifizierte die gleichen Y132F- und K143R-Substitutionen. Nach heterologer Expression dieser beiden Mutationen in S. cerivisiae waren die MHKs für Azole im Vergleich zu S. cerevisiae-Stämmen, die das Wildtyp-C. auris ERG11-Gen exprimieren, um das Zweifache erhöht (Healey et al., 2018).
2.1.1.3. ERG11-Überexpression
In C. albicans wurde eine Überexpression von ERG11 auch mit einer Resistenz gegen eine Azol-Behandlung in Verbindung gebracht.Die erhöhte Produktion von LD überwältigt die Fähigkeit des Antimykotikums, die Aktivität des Proteins zu hemmen, was dazu führt, dass das Protein trotz der medikamentösen Behandlung aktiv bleibt (Lopez-Ribot et al, 1998).
In C. auris zeigten Echtzeit-PCR-Experimente, dass es in Abwesenheit von Fluconazol keinen Unterschied in der ERG11-Expression zwischen Fluconazol-empfänglichen und Fluconazol-resistenten Stämmen gab (Chowdhary et al., 2018). In Gegenwart des Antimykotikums steigt die ERG11-Expression in resistenten Stämmen im Vergleich zu einer Kontrolle ohne Fluconazol an (Chowdhary et al., 2018). Die Wirkung dieses Medikaments auf die ERG11-Expression wurde jedoch nicht bei anfälligen Stämmen getestet. Daher konnte bisher nicht nachgewiesen werden, dass die bedingt erhöhte Expression von ERG11 in Gegenwart von Fluconazol spezifisch für resistente Stämme ist.
2.1.2. Mechanismen der Resistenz gegen Echinocandine
Beta(1,3)D-Glucan ist eine Schlüsselkomponente der Pilzzellwand und wird von den Genen FKS1 und FKS2 kodiert, zwei Untereinheiten der Beta(1,3)D-Glucan-Synthase. Echinocandine hemmen dieses Enzym, wodurch die Menge an Glucanen in der Zellwand verringert wird (Martins et al., 2011).
Bei C. albicans und anderen Candida-Spezies, die nicht zu den Cauris-Arten gehören, wurden mehrere Mutationen, die zu Echinocandin-Resistenz führen, in denselben beiden Regionen von FKS1 und FKS2 gefunden. Sie wurden daher als „Hot-Spots“ 1 und 2 (HS1 und HS2) bezeichnet. Im C. albicans FKS1-Gen befinden sich diese „Hot-Spots“ zwischen den Aminosäuren 641-649 und 1.345-1.365 (Park et al., 2005). Die Sequenzierung der entsprechenden Hot-Spot-Regionen von 38 C. auris-Stämmen führte zur Entdeckung einer S639F-Aminosäuresubstitution, die mit der Pan-Echinocandin-Resistenz korreliert ist: Sie war in allen vier resistenten Stämmen vorhanden und fehlte in den 34 anfälligen. Diese Position in C. auris FKS1 ist an die Position 645 (ebenfalls ein Serin) in C. albicans angeglichen (Chowdhary et al., 2018). Interessanterweise befindet sich diese Mutation in der Region, die mit dem HS1 von C. albicans FKS1 übereinstimmt. In anderen Studien wurden in Echinocandin-resistenten C. auris-Stämmen verschiedene Mutationen an der gleichen Stelle beobachtet: S639Y und S639P (Rhodes et al., 2018). Letztere wurde in einem Mausmodell als Ursache für Echinocandin-Resistenz in vivo bestätigt (Kordalewska et al., 2018).
FKS2 wurde ebenfalls in einer einzigen Kopie im Genom von C. auris gefunden (Sharma et al., 2016), aber unseres Wissens wurde in diesem Gen keine mit Echinocandin-Resistenz assoziierte Mutation gefunden.
2.1.3. Mechanismen der Resistenz gegen Polyene
Wie in Abbildung 1B gezeigt, sind mehrere C. auris-Stämme resistent gegen Amphotericin B (AMB). Bei den Candida-Arten wurden Veränderungen der Sterolzusammensetzung der Membran als Resistenzmechanismus hervorgehoben (Haynes et al., 1996; Nolte et al., 1997). Bei C. albicans wurde gezeigt, dass Mutationen in ERG 2, 3, 5, 6 oder 11 diese Wirkung haben (Arendrup und Patterson, 2017). Rhodes et al. untersuchten 27 C. auris-Isolate aus dem Vereinigten Königreich auf SNPs in diesen Genen in Stämmen, die eine verminderte Empfindlichkeit gegenüber AMB aufweisen. Es wurden jedoch keine Varianten gefunden, die diese Unterschiede in der Arzneimittelanfälligkeit erklären (Rhodes et al., 2018).
2.1.4. Mechanismen der Resistenz gegen Flucytosin (5-Fluorcytosin)
Flucytosin ist ein Nukleosidanalogon, das die Nukleinsäuresynthese hemmt. Nach dem Eintritt in die Zelle muss Flucytosin aktiviert werden, um eine antimykotische Wirkung zu entfalten. Für diese Aktivierung ist unter anderem das Protein erforderlich, das von dem Gen FUR1 Waldorf und Polak (1983) kodiert wird. Bei Candida-Arten, die nicht zu den auris-Arten gehören, wurde ein Zusammenhang zwischen Mutationen in FUR1 und Flucytosin-Resistenz nachgewiesen (Vandeputte et al., 2012).
Rhodes et al. sequenzierten einen Flucytosin-resistenten C. auris-Stamm und beobachteten eine F211I-Aminosäure-Substitution im FUR1-Gen (Rhodes et al., 2018). Diese spezifische Missense-Mutation hat kein bekanntes Äquivalent in anderen Candida-Arten, daher sind weitere Studien erforderlich, um festzustellen, ob diese Mutation die Ursache für die Resistenz gegen Flucytosin im getesteten C. auris-Stamm ist (Rhodes et al., 2018). Dieses Medikament wird jedoch weniger häufig eingesetzt als andere Antimykotika. Aus diesem Grund wurden weniger Studien durchgeführt, um die Resistenz von C. auris gegenüber diesem Wirkstoff besser zu verstehen.
Zusammenfassend lässt sich sagen, dass einige Resistenzmechanismen, die zuvor bei Candida-Arten wie C. glabrata und C. albicans entdeckt wurden, auch bei C. auris zu beobachten waren. Sie sind in der letzten Spalte der Tabelle 1 aufgeführt. Die antimykotische Resistenz von C. auris lässt sich jedoch nur teilweise durch diese zuvor beschriebenen Resistenzmechanismen erklären. Um schließlich neue therapeutische Wirkstoffe zu entwickeln und generell die Patientenversorgung zu verbessern, sind weitere Studien erforderlich, um die zugrunde liegenden Mechanismen besser zu verstehen, die zu den außergewöhnlich hohen MHK-Bereichen einiger C. auris-Stämme (siehe Abbildung 1B).
2.2. Biofilm: Eine antimykotische resistente Lebensweise
Es wurde gezeigt, dass die MHKs für mehrere Antimykotika in sessilen Zellen von C. auris höher sind (bis zu 4-, 20- bzw. 60-fach für Voriconazol, Amphotericin B und Micafungin) im Vergleich zu planktonischen Zellen (Sherry et al., 2017). In ähnlicher Weise wurde gezeigt, dass die minimalen Biofilm-Eradikationskonzentrationen 512-fach höher sind als die MHKs für Echinocandine und Azole (Romera et al., 2019). Dieses Phänomen wurde bereits für C. albicans nachgewiesen (Hawser und Douglas, 1995). Die molekularen Mechanismen, die für diese erhöhten MHKs bei C. auris verantwortlich sind, sind noch weitgehend unbekannt, aber einige Studien haben bereits einige Einblicke geliefert.
MSF- und ABC-Effluxpumpen-kodierende Gene wurden in sessilen Zellen im Vergleich zu planktonischen Zellen hochreguliert (2- bis 4-fach) (Kean et al., 2018a). Gleichzeitig war die Aktivität der entsprechenden Proteine um das 2-fache erhöht. Bei Behandlung mit Effluxpumpen-Inhibitoren war die antimykotische Empfindlichkeit der sessilen Zellen nach 12 Stunden jedoch um das 4- bis 16-fache erhöht. Dies deutet darauf hin, dass Effluxpumpen eine wichtige Rolle bei der Resistenz dieses Zelltyps gegen Antimykotika spielen (Kean et al., 2018a).
Bei C. albicans ist bekannt, dass die Exopolymermatrix unspezifisch an alle Klassen von Antimykotika bindet und diese außerhalb der Zellen sequestriert (Nett et al., 2010). Da Candida spp. ein gemeinsames Kernpolysaccharidprofil aufweisen, wurde vermutet, dass dieser Mechanismus auch in C. auris konserviert sein könnte (Kean et al., 2018a). Dies wurde später in In-vivo- und In-vitro-Tests bestätigt (Dominguez et al., 2019).
Nosokomiale Persistenz
Die Fähigkeit von C. auris, sich auf Oberflächen zu kolonisieren und zu persistieren, ist außergewöhnlich: Es kann im Vergleich zu C. albicans länger auf feuchten Oberflächen persistieren (Piedrahita et al., 2017). Er hat auch eine verlängerte Stoffwechselaktivität auf Oberflächen, ähnlich wie C. parapsilosis, ein bekannter Haut- und Kunststoffbesiedler (Piedrahita et al., 2017; Welsh et al., 2017).
Das erhöhte Überleben von C. auris auf Oberflächen wurde in zwei Studien untersucht. Die erste behauptete, dass es mit einer erhöhten Umweltstressresistenz gegenüber Temperatur und anderen Stressoren zusammenhängen könnte (Kean et al., 2018b). Die zweite stellt die Hypothese auf, dass dies auf die Bildung eines Biofilms zurückzuführen sein könnte. Dies muss jedoch noch bewiesen werden, da bisher kein biofilmbildendes Isolat von Umweltoberflächen gewonnen wurde (Ku et al., 2018). Es ist anzumerken, dass sich diese beiden Möglichkeiten nicht gegenseitig ausschließen und dass die Biofilmbildung sogar die Ursache für die erhöhte Stressresistenz sein könnte (Ku et al., 2018).
Die Persistenz von C. auris in nosokomialen Umgebungen wird durch die Resistenz dieses Erregers gegenüber Desinfektionsverfahren verstärkt. Getestet wurde die Wirksamkeit von NaOCl und Peressigsäure auf Oberflächen aus Edelstahl, Polymer (Polyester-Deckgläser) und Zellulose. Beide Desinfektionsmittel zeigten eine signifikante Wirksamkeit bei der Abtötung von C. auris-Zellen auf allen Oberflächen. Nach der Anwendung von NaOCl auf nicht porösen Oberflächen (Edelstahl und Polyester-Deckgläser) blieben jedoch einige lebensfähige Zellen zurück. Tatsächlich waren höhere Konzentrationen dieses Desinfektionsmittels in Verbindung mit längeren Einwirkungszeiten erforderlich, um das erneute Wachstum zu verringern, aber selbst dann konnten sie den Erreger nicht vollständig abtöten. So reichte beispielsweise eine NaOCl-Konzentration von 10.000 Teilen pro Million (ppm) in Verbindung mit einer 5-minütigen Einwirkungszeit auf rostfreiem Stahl aus, um eine deutliche Reduzierung der Kolonien zu beobachten. Nach einer erneuten Beimpfung auf einem reichhaltigen Medium wurde jedoch ein erhebliches Nachwachsen beobachtet (Kean et al., 2018b).
Bei Peressigsäure, die ein hochwirksames Desinfektionsmittel ist, war dieses Nachwachsen oberflächenabhängig, da bei Polymer im Gegensatz zu Edelstahl kein Nachwachsen beobachtet wurde. Sie führten dieselben Experimente mit C. glabrata und C. albicans durch und kamen zu ähnlichen Ergebnissen (Kean et al., 2018b).
Das zeigt, dass hochwirksame Desinfektionsverfahren für eine vollständige Reinigung der Oberflächen möglicherweise nicht ausreichen und dass die bereits etablierten Richtlinien für die Flächendesinfektion an diese Candida-Spezies angepasst werden müssen (Kean et al., 2018b). Bei der Umsetzung dieser Desinfektionsverfahren sollten Faktoren wie die Art der Flächen und die Einwirkungszeiten berücksichtigt werden (Kean et al., 2018b; Ku et al., 2018), nicht nur die Kosten und die einfache Anwendung, wie es oft der Fall ist (Ku et al., 2018).
Basierend auf den Ergebnissen von zwei Studien (Cadnum et al., 2017; Rutala et al., 2019) empfiehlt die CDC die Verwendung von Desinfektionsmitteln, die gegen Clostridioides difficile-Sporen wirksam sind. Falls dies nicht möglich ist, empfehlen sie Alternativen wie Wasserstoffperoxid 0,5-1,4 % oder quaternäre Ammoniumverbindungen mit Isopropylalkohol und/oder Ethylalkohol2. Schließlich könnte auch ultraviolettes C-Licht ein interessanter Kandidat für die Oberflächendesinfektion sein. Es erwies sich als äußerst wirksam bei der Abtötung von C. auris-Kolonien, sofern die Bestrahlungszeit und der Abstand ausreichend waren. Vor der Anwendung solcher Methoden in Krankenhäusern sind jedoch weitere Studien erforderlich (de Groot et al., 2019).
Schlussfolgerung
Die pathogenen und virulenten Fähigkeiten von C. auris sind zutiefst beunruhigend. Vor allem, da die Ausbrüche unabhängig voneinander in verschiedenen Regionen der Welt auftraten und die assoziierten Isolate eine verringerte Empfindlichkeit gegenüber den am häufigsten verwendeten antimykotischen Arzneimitteln zur Behandlung von Patienten aufwiesen (siehe Abbildung 1).
Um die verringerte antimykotische Empfindlichkeit bei C. auris zu erklären, nutzten die Forscher das bereits vorhandene umfangreiche Wissen über andere Candida-Arten, insbesondere C. albicans. Als Ergebnis wurden einige der Resistenzmechanismen von C. auris hervorgehoben (siehe Tabelle 1). Genauer gesagt wurde nachgewiesen, dass zwei Punktmutationen in ERG11 und die Überexpression des ABC-Transporters Cdr1 die Fluconazolempfindlichkeit verringern. Darüber hinaus wurde gezeigt, dass eine Aminosäuresubstitution in FKS1 die Empfindlichkeit von C. auris gegenüber Echinocandinen verringert, die normalerweise das Mittel der Wahl zur Behandlung dieses Erregers sind (Bidaud et al., 2018). Schließlich wurde in einem Flucytosin-resistenten C. auris-Stamm eine Mutation in FUR1 identifiziert, deren Ursache für die verminderte Empfindlichkeit jedoch noch nicht nachgewiesen werden konnte. Es wird angenommen, dass Biofilm per se ein Resistenzmechanismus ist, da gezeigt wurde, dass er die Medikamentenempfindlichkeit verringert. Es sind jedoch noch weitere Forschungsarbeiten erforderlich, um die ganze Vielfalt der Prozesse zu beschreiben, die in dieser Wachstumsform ablaufen. Es ist wichtig, darauf hinzuweisen, dass der Biofilm eine komplexe Struktur ist, die selbst bei bekannten Arten wie C. albicans kaum verstanden wird und schwer zu erforschen ist.
Die Resistenzeigenschaften von C. auris sind mit der Neigung verbunden, nosokomiale Infektionen zu verursachen. Seine Fähigkeit, an einer Vielzahl von nosokomialen Oberflächen zu haften und die Desinfektion zu überleben, sind entscheidende Ausbreitungsfaktoren. Das Vorhandensein mehrerer Arten von Oberflächen stellt für klinische Einrichtungen eine Herausforderung dar, da die Standarddesinfektionsverfahren ineffizient und nicht auf die einzelnen Arten von Oberflächen abgestimmt sind. Dieses Problem ergibt sich auch aus der unzureichenden Forschung zu diesem Thema. Darüber hinaus würde ein besseres Verständnis der Mechanismen, die der Resistenz von C. auris gegen Desinfektionsmittel zugrunde liegen, dazu beitragen, die Hygieneprotokolle zu verbessern und künftige nosokomiale Ausbrüche zu vermeiden.
Beiträge der Autoren
FC, AG und LJ waren zu gleichen Teilen an der Abfassung dieser Übersichtsarbeit beteiligt. AC überwachte und beteiligte sich an der Redaktion dieser Übersichtsarbeit.
Interessenkonflikt
Die Autoren erklären, dass die Forschung ohne jegliche kommerzielle oder finanzielle Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Wir danken Clara Heiman als englischer Muttersprachlerin für die Überarbeitung unseres Manuskripts.
Fußnoten
1. ^CDC (2019). Antifungal Susceptibility Testing and Interpretation. Online verfügbar unter: https://www.cdc.gov/fungal/candida-auris/c-auris-antifungal.html (Zugriff am 29. April 2019).
2. ^CDC (2018). Infektionsprävention und -kontrolle für Candida auris. Online verfügbar unter: https://www.cdc.gov/fungal/candida-auris/c-auris-infection-control.html (Zugriff am 12. November 2019).
Abdalhamid, B., Almaghrabi, R., Althawadi, S., und Omrani, A. (2018). Erster Bericht über eine Candida auris-Infektion aus Saudi-Arabien. J. Infect. Public Health 11, 598-599. doi: 10.1016/j.jiph.2018.05.010
PubMed Abstract | CrossRef Full Text | Google Scholar
Adam, R. D., Revathi, G., Okinda, N., Fontaine, M., Shah, J., Kagotho, E., et al. (2019). Analyse der Candida auris Fungämie in einer einzigen Einrichtung in Kenia. Int. J. Infect. Dis. 85, 182-187. doi: 10.1016/j.ijid.2019.06.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Adams, E., Quinn, M., Tsay, S., Poirot, E., Chaturvedi, S., Southwick, K., et al. (2018). Candida auris in Gesundheitseinrichtungen, New York, USA, 2013-2017. Emerg. Infect. Dis. 24, 1816-1824. doi: 10.3201/eid2410.180649
CrossRef Full Text | Google Scholar
Arendrup, M. C., and Patterson, T. F. (2017). Multimittelresistente Candida: Epidemiologie, molekulare Mechanismen und Behandlung. J. Infect. Dis. 216, S445-S451. doi: 10.1093/infdis/jix131
PubMed Abstract | CrossRef Full Text | Google Scholar
Barantsevich, N. E., Orlova, O. E., Shlyakhto, E. V., Johnson, E. M., Woodford, N., Lass-Floerl, C., et al. (2019). Auftreten von Candida auris in Russland. J. Hosp. Infect. 102, 445-448. doi: 10.1016/j.jhin.2019.02.021
PubMed Abstract | CrossRef Full Text | Google Scholar
Belkin, A., Gazit, Z., Keller, N., Ben-Ami, R., Wieder-Finesod, A., Novikov, A., et al. (2018). Candida auris-Infektion, die zu einer nosokomialen Übertragung führt, Israel, 2017. Emerg. Infect. Dis. 24, 801-804. doi: 10.3201/eid2404.171715
PubMed Abstract | Riff Full Text | Google Scholar
Ben-Ami, R., Berman, J., Novikov, A., Bash, E., Shachor-Meyouhas, Y., Zakin, S., et al. (2017). Multidrug-resistant Candida haemulonii and C. auris, Tel Aviv, Israel. Emerg. Infect. Dis. 23, 195-203. doi: 10.3201/eid2302.161486
PubMed Abstract | CrossRef Full Text | Google Scholar
Bidaud, A. L., Chowdhary, A., and Dannaoui, E. (2018). Candida auris: ein aufstrebender arzneimittelresistenter Hefepilz – Ein Mini-Review. J. Mycol. Méd. 28, 568-573. doi: 10.1016/j.mycmed.2018.06.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Biswal, M., Rudramurthy, S. M., Jain, N., Shamanth, A. S., Sharma, D., Jain, K., et al. (2017). Kontrolle eines möglichen Ausbruchs einer Candida auris-Infektion: Lektionen aus mehreren Interventionen gelernt. J. Hosp. Infect. 97, 363-370. doi: 10.1016/j.jhin.2017.09.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Borman, A. M., Szekely, A., and Johnson, E. M. (2016). Comparative pathogenicity of United Kingdom isolates of the emerging pathogen Candida auris and other key pathogenic candida species. mSphere 1:e00189-16. doi: 10.1128/mSphere.00189-16
CrossRef Full Text | Google Scholar
Cadnum, J. L., Shaikh, A. A., Piedrahita, C. T., Sankar, T., Jencson, A. L., Larkin, E. L., et al. (2017). Effektivität von Desinfektionsmitteln gegen Candida auris und andere Candida-Arten. Infect. Control Hosp. Epidemiol. 38, 1240-1243. doi: 10.1017/ice.2017.162
PubMed Abstract | CrossRef Full Text | Google Scholar
Chatterjee, S., Alampalli, S. V., Nageshan, R. K., Chettiar, S. T., Joshi, S., and Tatu, U. S. (2015). Draft genome of a commonly misdiagnosed multidrug resistant pathogen Candida auris. BMC Genomics 16:686. doi: 10.1186/s12864-015-1863-z
PubMed Abstract | CrossRef Full Text | Google Scholar
Chow, N. A., Gade, L., Tsay, S. V., Forsberg, K., Greenko, J. A., Southwick, K. L., et al. (2018). Multiple Einschleppung und anschließende Übertragung von multiresistenten Candida auris in den USA: eine molekulare epidemiologische Untersuchung. Lancet Infect. Dis. 18, 1377-1384. doi: 10.1016/S1473-3099(18)30597-8
PubMed Abstract | CrossRef Full Text | Google Scholar
Chowdhary, A., Prakash, A., Sharma, C., Kordalewska, M., Kumar, A., Sarma, S., et al. (2018). Eine multizentrische Studie zu Antimykotika-Empfindlichkeitsmustern unter 350 Candida auris-Isolaten (2009-17) in Indien: Rolle der ERG11- und FKS1-Gene bei Azol- und Echinocandin-Resistenz. J. Antimicrob. Chemother. 73, 891-899. doi: 10.1093/jac/dkx480
PubMed Abstract | CrossRef Full Text | Google Scholar
de Groot, T., Chowdhary, A., Meis, J. F., and Voss, A. (2019). Abtötung von Candida auris durch UV-C: Bedeutung von Expositionszeit und Abstand. Mycoses 62, 408-412. doi: 10.1111/myc.12903
PubMed Abstract | CrossRef Full Text | Google Scholar
Desoubeaux, G., Bailly, E., Guillaume, C., De Kyvon, M.-A., Tellier, A.-C., Morange, V., et al. (2018). Candida auris in modernen Mykologielaboren: einige praktische Tricks, um ihn zuverlässig zu identifizieren, wie eine aktuelle französische Erfahrung zeigt. J. Mycol. Med. 28, 407-410. doi: 10.1016/j.mycmed.2018.02.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Dominguez, E. G., Zarnowski, R., Choy, H. L., Zhao, M., Sanchez, H., Nett, J. E., et al. (2019). Conserved role for biofilm matrix polysaccharides in Candida auris drug resistance. mSphere 4:e00680-18. doi: 10.1128/mSphereDirect.00680-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Escandón, P., Chow, N. A., Caceres, D. H., Gade, L., Berkow, E. L., Armstrong, P., et al. (2019). Molekulare Epidemiologie von Candida auris in Kolumbien zeigt eine hochgradig verwandte, landesweite Kolonisation mit regionalen Mustern der Amphotericin-B-Resistenz. Clin. Infect. Dis. 68, 15-21. doi: 10.1093/cid/ciy411
CrossRef Full Text | Google Scholar
Eyre, D. W., Sheppard, A. E., Madder, H., Moir, I., Moroney, R., Quan, T. P., et al. (2018). Ein Ausbruch von Candida auris und seine Kontrolle in einer Intensivpflegeeinrichtung. N. Engl. J. Med. 379, 1322-1331. doi: 10.1056/NEJMoa1714373
PubMed Abstract | CrossRef Full Text | Google Scholar
Fakhim, H., Vaezi, A., Dannaoui, E., Chowdhary, A., Nasiry, D., Faeli, L., et al. (2018). Vergleichende Virulenz von Candida auris mit Candida haemulonii, Candida glabrata und Candida albicans in einem murinen Modell. Mycoses 61, 377-382. doi: 10.1111/myc.12754
PubMed Abstract | CrossRef Full Text | Google Scholar
Hawser, S. P., and Douglas, L. J. (1995). Resistenz von Candida albicans-Biofilmen gegen Antimykotika in vitro. Antimicrob. Agents Chemother. 39, 2128-2131. doi: 10.1128/AAC.39.9.2128
PubMed Abstract | CrossRef Full Text | Google Scholar
Haynes, M. P., Chong, P. L.-G., Buckley, H. R., and Pieringer, R. A. (1996). Fluoreszenzstudien über die molekulare Wirkung von Amphotericin B auf anfällige und resistente Pilzzellen. Biochemistry 35, 7983-7992. doi: 10.1021/bi952910c
PubMed Abstract | CrossRef Full Text | Google Scholar
Healey, K. R., Kordalewska, M., Ortigosa, C. J., Singh, A., Berrío, I., Chowdhary, A., et al. (2018). Begrenzte ERG11-Mutationen, die in Isolaten von Candida auris identifiziert wurden, tragen direkt zu einer reduzierten Azol-Empfindlichkeit bei. Antimicrob. Agents Chemother. 62, e01427-18. doi: 10.1128/AAC.01427-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Jackson, B. R., Chow, N., Forsberg, K., Litvintseva, A. P., Lockhart, S. R., Welsh, R., et al. (2019). On the origins of a species: what might explain the rise of Candida auris? J. Fungi 5:E58. doi: 10.3390/jof5030058
PubMed Abstract | CrossRef Full Text | Google Scholar
Kean, R., Delaney, C., Sherry, L., Borman, A., Johnson, E. M., Richardson, M. D., et al. (2018a). Transcriptome assembly and profiling of Candida auris reveals novel insights into biofilm-mediated resistance. mSphere 3:e00334-18. doi: 10.1128/mSphere.00334-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Kean, R., Sherry, L., Townsend, E., McKloud, E., Short, B., Akinbobola, A., et al. (2018b). Herausforderungen bei der Flächendesinfektion von Candida auris: eine In-vitro-Studie. J. Hosp. Infect. 98, 433-436. doi: 10.1016/j.jhin.2017.11.015
PubMed Abstract | CrossRef Full Text | Google Scholar
Kenters, N., Kiernan, M., Chowdhary, A., Denning, D. W., Pemán, J., Saris, K., et al. (2019). Kontrolle von Candida auris in Gesundheitseinrichtungen. Outcome of an ISAC expert meeting. Int. J. Antimicrob. Agents 54, 400-406. doi: 10.1016/j.ijantimicag.2019.08.013
CrossRef Full Text | Google Scholar
Kordalewska, M., Lee, A., Park, S., Berrio, I., Chowdhary, A., Zhao, Y., et al. (2018). Understanding echinocandin resistance in the emerging pathogen Candida auris. Antimicrob. Agents Chemother. 62:e00238-18. doi: 10.1128/AAC.00238-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Krishnasamy, L., Krishnakumar, S., Kumaramanickavel, G., and Saikumar, C. (2018). Molekulare Mechanismen der antimykotischen Arzneimittelresistenz bei Candida-Arten. J. Clin. Diagn. Res. 12, DE01-DE06. doi: 10.7860/JCDR/2018/36218.11961
CrossRef Full Text | Google Scholar
Ku, T. S. N., Walraven, C. J., and Lee, S. A. (2018). Candida auris: Desinfektionsmittel und Implikationen für die Infektionskontrolle. Front. Microbiol. 9:726. doi: 10.3389/fmicb.2018.00726
CrossRef Full Text | Google Scholar
Leach, L., Russell, A., Zhu, Y., Chaturvedi, S., and Chaturvedi, V. (2019). A rapid and automated sample-to-result Candida auris real-time PCR assay for high-throughput testing of surveillance samples with BD MAX™ open system. J. Clin. Microbiol. 57:e00630-19. doi: 10.1128/JCM.00630-19
CrossRef Full Text | Google Scholar
Lesho, E. P., Bronstein, M. Z., McGann, P., Stam, J., Kwak, Y., Maybank, R., et al. (2018). Einschleppung, Eindämmung und genomische Epidemiologie von Candida auris in einem großen Lehrkrankenhaus. Infect. Control Hosp. Epidemiol. 39, 53-57. doi: 10.1017/ice.2017.231
PubMed Abstract | CrossRef Full Text | Google Scholar
Lockhart, S. R., Etienne, K. A., Vallabhaneni, S., Farooqi, J., Chowdhary, A., Govender, N. P., et al. (2017). Gleichzeitiges Auftreten von multiresistenten Candida auris auf 3 Kontinenten bestätigt durch Ganzgenomsequenzierung und epidemiologische Analysen. Clin. Infect. Dis. 64, 134-140. doi: 10.1093/cid/ciw691
PubMed Abstract | CrossRef Full Text | Google Scholar
Lopez-Ribot, J. L., McAtee, R. K., Lee, L. N., Kirkpatrick, W. R., White, T. C., Sanglard, D., et al. (1998). Unterschiedliche Muster der Genexpression im Zusammenhang mit der Entwicklung einer Fluconazol-Resistenz bei seriellen Candida albicans-Isolaten von mit dem Humanen Immundefizienz-Virus infizierten Patienten mit oropharyngealer Candidose. Antimicrob. Agents Chemother. 42, 2932-2937. doi: 10.1128/AAC.42.11.2932
PubMed Abstract | CrossRef Full Text | Google Scholar
Martins, I. M., Cortés, J. C. G., Muñoz, J., Moreno, M. B., Ramos, M., Clemente-Ramos, J. A., et al. (2011). Unterschiedliche Aktivitäten von drei Familien spezifischer Beta(1,3)-Glucan-Synthase-Inhibitoren in Wildtyp- und resistenten Stämmen der Spalthefe. J. Biol. Chem. 286, 3484-3496. doi: 10.1074/jbc.M110.174300
PubMed Abstract | CrossRef Full Text | Google Scholar
Mayor, S. (2018). Candida auris outbreak was linked to reusable axillary temperature probes, study finds. BMJ 363:k4133. doi: 10.1136/bmj.k4133
PubMed Abstract | CrossRef Full Text | Google Scholar
Morschhäuser, J., Barker, K. S., Liu, T. T., Bla-Warmuth, J., Homayouni, R., and Rogers, P. D. (2007). Der Transkriptionsfaktor Mrr1p kontrolliert die Expression der MDR1-Effluxpumpe und vermittelt die Multidrug-Resistenz von Candida albicans. PLoS Pathog. 3:e164. doi: 10.1371/journal.ppat.0030164
PubMed Abstract | CrossRef Full Text | Google Scholar
Nett, J. E., Crawford, K., Marchillo, K., and Andes, D. R. (2010). Die Rolle von Fks1p und Matrix-Glucan bei der Resistenz von Candida albicans-Biofilmen gegen ein Echinocandin, Pyrimidin und Polyen. Antimicrob. Agents Chemother. 54, 3505-3508. doi: 10.1128/AAC.00227-10
PubMed Abstract | CrossRef Full Text | Google Scholar
Nolte, F. S., Parkinson, T., Falconer, D. J., Dix, S., Williams, J., Gilmore, C., et al. (1997). Isolierung und Charakterisierung von Fluconazol- und Amphotericin B-resistenten Candida albicans aus dem Blut von zwei Patienten mit Leukämie. Antimicrob. Agents Chemother. 41, 196-199. doi: 10.1128/AAC.41.1.196
PubMed Abstract | CrossRef Full Text | Google Scholar
O’Connor, C., Bicanic, T., Dave, J., Evans, T. J., Moxey, P., Adamu, U., et al. (2019). Ausbruch von Candida auris auf einer Gefäßstation – die unerwartete Ankunft eines erwarteten Erregers. J. Hosp. Infect. 103, 106-108. doi: 10.1016/j.jhin.2019.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Osei Sekyere, J. (2018). Candida auris: a systematic review and meta-analysis of current updates on an emerging multidrug-resistant pathogen. MicrobiologyOpen 7:e00578. doi: 10.1002/mbo3.578
PubMed Abstract | CrossRef Full Text | Google Scholar
Park, J. Y., Bradley, N., Brooks, S., Burney, S., and Wassner, C. (2019). Behandlung von Patienten mit Candida auris-Fungämie in einem Gemeinschaftskrankenhaus, Brooklyn, New York, USA, 2016-20181. Emerg. Infect. Dis. 25, 601-602. doi: 10.3201/eid2503.180927
PubMed Abstract | CrossRef Full Text | Google Scholar
Park, S., Kelly, R., Kahn, J. N., Robles, J., Hsu, M.-J., Register, E., et al. (2005). Spezifische Substitutionen im Echinocandin-Target Fks1p sind für die verringerte Empfindlichkeit seltener Labor- und klinischer Isolate von Candida sp. verantwortlich. Antimicrob. Agents Chemother. 49, 3264-3273. doi: 10.1128/AAC.49.8.3264-3273.2005
CrossRef Full Text | Google Scholar
Parra-Giraldo, C. M., Valderrama, S. L., Cortes-Fraile, G., Garzón, J. R., Ariza, B. E., Morio, F., et al. (2018). Erster Bericht über sporadische Fälle von Candida auris in Kolumbien. Int. J. Infect. Dis. 69, 63-67. doi: 10.1016/j.ijid.2018.01.034
PubMed Abstract | CrossRef Full Text | Google Scholar
Pemán, J., Cantón, E., and Espinel-Ingroff, A. (2009). Antifungal drug resistance mechanisms. Expert Rev. Anti Infect. Ther. 7, 453-460. doi: 10.1586/eri.09.18
PubMed Abstract | CrossRef Full Text | Google Scholar
Piedrahita, C. T., Cadnum, J. L., Jencson, A. L., Shaikh, A. A., Ghannoum, M. A., and Donskey, C. J. (2017). Umgebungsoberflächen in Gesundheitseinrichtungen sind eine potenzielle Quelle für die Übertragung von Candida auris und anderen Candida-Arten. Infect. Control Hosp. Epidemiol. 38, 1107-1109. doi: 10.1017/ice.2017.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Rhodes, J., Abdolrasouli, A., Farrer, R. A., Cuomo, C. A., Aanensen, D. M., Armstrong-James, D., et al. (2018). Genomic epidemiology of the UK outbreak of the emerging human fungal pathogen Candida auris. Emerg. Microbes Infect. 7:43. doi: 10.1101/201343
CrossRef Full Text | Google Scholar
Romera, D., Aguilera-Correa, J. J., Gadea, I., Viñuela-Sandoval, L., García-Rodríguez, J., and Esteban, J. (2019). Candida auris: ein Vergleich zwischen planktonischer und Biofilm-Empfindlichkeit gegenüber Antimykotika. J. Med. Microbiol. 68, 1353-1358. doi: 10.1099/jmm.0.001036
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A., Martínez, H., Moret, A. M., Calabuig, E., Tasias, M., Alastruey-Izquierdo, A., et al. (2019a). Erkennung und Behandlung von Candida auris in einer Ausbruchssituation: Risikofaktoren für die Entwicklung einer Kolonisierung und Candidämie durch diese neue Spezies bei kritisch kranken Patienten. Expert Rev. Anti Infect. Ther. 17, 295-305. doi: 10.1080/14787210.2019.1592675
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A., Moret, A. M., Tasias-Pitarch, M., Aleixandre-López, A. I., Martínez-Morel, H., Calabuig, E., et al. (2018). Ein Ausbruch von Candida auris mit verlängerter Kolonisation und Candidämie in einem europäischen Krankenhaus der Tertiärversorgung. Mycoses 61, 498-505. doi: 10.1111/myc.12781
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A. C., Cantón, E., Fernández-Rivero, M. E., Ramírez, P., and Pemán, J. (2019b). Ausbruch von Candida auris in Spanien: ein Vergleich der antimykotischen Aktivität durch drei Methoden mit veröffentlichten Daten. Int. J. Antimicrob. Agents 53, 541-546. doi: 10.1016/j.ijantimicag.2019.02.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Rutala, W. A., Kanamori, H., Gergen, M. F., Sickbert-Bennett, E. E., and Weber, D. J. (2019). Anfälligkeit von Candida auris und Candida albicans gegenüber 21 in Gesundheitseinrichtungen verwendeten Germiziden. Infect. Control Hosp. Epidemiol. 40, 380-382. doi: 10.1017/ice.2019.1
PubMed Abstract | CrossRef Full Text | Google Scholar
Rybak, J. M., Doorley, L. A., Nishimoto, A. T., Barker, K. S., Palmer, G. E., and Rogers, P. D. (2019). Aufhebung der Triazolresistenz durch Deletion von CDR1 in einem klinischen Isolat von Candida auris. Antimicrob. Agents Chemother. 63, e00057-19. doi: 10.1128/AAC.00057-19
PubMed Abstract | CrossRef Full Text | Google Scholar
Sana, F., Hussain, W., Zaman, G., Satti, L., Khurshid, U., and Khadim, M. T. (2019). Bericht über einen Ausbruch von Candida auris in Pakistan: eine Erfolgsgeschichte der Infektionskontrolle auf der Intensivstation eines Krankenhauses der Tertiärversorgung. J. Hosp. Infect. 103, 108-110. doi: 10.1016/j.jhin.2019.06.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Sanglard, D., Coste, A., and Ferrari, S. (2009). Mechanismen der Antimykotika-Resistenz bei Pilzpathogenen aus der Perspektive der transkriptionellen Genregulation. FEMS Yeast Res. 9, 1029-1050. doi: 10.1111/j.1567-1364.2009.00578.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Sanglard, D., Ischer, F., Koymans, L., and Bille, J. (1998). Aminosäure-Substitutionen im Cytochrom P-450 Lanosterol 14-Demethylase (CYP51a1) von Azol-resistenten klinischen Isolaten von Candida albicans tragen zur Resistenz gegen Azol-Antimykotika bei. Antimicrob. Agents Chemother. 42, 241-253.
Google Scholar
Sanglard, D., Kuchler, K., Ischer, F., Pagani, J. L., Monod, M., and Bille, J. (1995). Mechanismen der Resistenz gegen Azol-Antimykotika in Candida albicans-Isolaten von AIDS-Patienten beinhalten spezifische Multidrug-Transporter. Antimicrob. Agents Chemother. 39, 2378-2386. doi: 10.1128/AAC.39.11.2378
PubMed Abstract | CrossRef Full Text | Google Scholar
Sayeed, M. A., Farooqi, J., Jabeen, K., Awan, S., and Mahmood, S. F. (2019). Klinisches Spektrum und Faktoren, die das Ergebnis von Candida auris beeinflussen: eine Studie an einem einzigen Zentrum in Pakistan. BMC Infect. Dis. 19:384. doi: 10.1186/s12879-019-3999-y
PubMed Abstract | CrossRef Full Text | Google Scholar
Schelenz, S., Hagen, F., Rhodes, J. L., Abdolrasouli, A., Chowdhary, A., Hall, A., et al. (2016). Erster Krankenhausausbruch des weltweit auftretenden Candida auris in einem europäischen Krankenhaus. Antimicrob. Resist. Infect. Control 5:35. doi: 10.1186/s13756-016-0132-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Schuetzer-Muehlbauer, M., Willinger, B., Egner, R., Ecker, G., and Kuchler, K. (2003). Umkehrung der antimykotischen Resistenz durch ABC-Effluxpumpen von Candida albicans, die funktionell in Hefe exprimiert werden. Int. J. Antimicrob. Agents 22, 291-300. doi: 10.1016/S0924-8579(03)00213-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Sexton, D. J., Kordalewska, M., Bentz, M. L., Welsh, R. M., Perlin, D. S., und Litvintseva, A. P. (2018). Direct detection of emergent fungal pathogen Candida auris in clinical skin swabs by SYBR green-based quantitative PCR assay. J. Clin. Microbiol. 56:e01337-18. doi: 10.1128/JCM.01337-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sharma, C., Kumar, N., Pandey, R., Meis, J. F., and Chowdhary, A. (2016). Whole Genome Sequencing of emerging multidrug resistant Candida auris isolates in India demonstrates low genetic variation. New Microbes New Infect. 13, 77-82. doi: 10.1016/j.nmni.2016.07.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Sherry, L., Ramage, G., Kean, R., Borman, A., Johnson, E. M., Richardson, M. D., et al. (2017). Biofilmbildende Fähigkeit von hochvirulentem, multiresistentem Candida auris. Emerg. Infect. Dis. 23, 328-331. doi: 10.3201/eid2302.161320
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandeputte, P., Ferrari, S., and Coste, A. T. (2012). Pilzresistenz und neue Strategien zur Bekämpfung von Pilzinfektionen. Int. J. Microbiol. 2012:713687. doi: 10.1155/2012/713687
PubMed Abstract | CrossRef Full Text | Google Scholar
Waldorf, A. R., and Polak, A. (1983). Wirkmechanismen von 5-Fluorocytosin. Antimicrob. Agents Chemother. 23, 79-85. doi: 10.1128/AAC.23.1.79
PubMed Abstract | CrossRef Full Text | Google Scholar
Wang, X., Bing, J., Zheng, Q., Zhang, F., Liu, J., Yue, H., et al. (2018). Das erste Isolat von Candida auris in China: klinische und biologische Aspekte. Emerging Microbes & Infections 7, 1-9. doi: 10.1038/s41426-018-0095-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Welsh, R. M., Bentz, M. L., Shams, A., Houston, H., Lyons, A., Rose, L. J., et al. (2017). Überleben, Persistenz und Isolierung des neuen multiresistenten pathogenen Hefepilzes Candida auris auf einer Kunststoffoberfläche im Gesundheitswesen. J. Clin. Microbiol. 55, 2996-3005. doi: 10.1128/JCM.00921-17
PubMed Abstract | CrossRef Full Text | Google Scholar
Leave a Reply