Frontiers in Microbiology
Introduction
Na última década, uma espécie emergente de Candida nosocomial tem causado casos graves de candidemia e outros tipos de infecções fúngicas. Surtos do patógeno responsável, nomeadamente Candida auris, têm sido observados em todo o mundo (ver Figura 1A) (Lockhart et al., 2017).
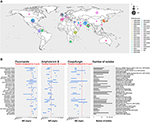
Figura 1. (A) Mapa representando o número de isolados por surto desde 1996. O primeiro ano do surto é de cor marrom e o último ano do surto é em rosa. O tamanho do círculo é proporcional ao número de isolados relatados para cada surto. A pesquisa de surtos foi realizada recuperando dados de três revisões principais (Osei Sekyere, 2018; Jackson et al., 2019; Kenters et al., 2019) e complementada por uma pesquisa pubmed usando a consulta “Candida” E “auris” E “surto” como palavras-chave. Os resultados foram limitados no tempo, entre 21/07/2017, o último estudo da revisão sistemática de Osei Sekyere e 31/08/2019, quando foi realizada a pesquisa bibliográfica. Estudos não presentes na revisão por Osei Sekyere: (Biswal et al., 2017; Abdalhamid et al., 2018; Adams et al., 2018; Belkin et al., 2018; Chow et al., 2018; Desoubeaux et al., 2018; Lesho et al., 2018; Mayor, 2018; Parra-Giraldo et al., 2018; Ruiz-Gaitán et al, 2018, 2019a,b; Sexton et al., 2018; Adam et al., 2019; Barantsevich et al., 2019; Escandón et al., 2019; Leach et al., 2019; O’Connor et al., 2019; Park et al., 2019; Sana et al., 2019; Sayeed et al., 2019). (B) Os intervalos MIC para três antifúngicos medidos em C. auris isolados de diferentes surtos ao redor do mundo. A linha pontilhada vermelha representa o ponto de quebra provisório proposto pelo CDC. IPSV é um acrônimo para “Índia, Paquistão, África do Sul e Venezuela”, já que as cepas desses países foram analisadas no mesmo estudo. A pesquisa de ruptura foi realizada de forma idêntica à (A). Somente foram traçados surtos com >1 isolados. Estudos citados por Osei Sekyere são sinalizados com e estudos citados por Kenters et al. com .
Sequenciamento de todo o genoma determinou a presença de quatro clades específicos dentro da espécie C. auris: Ásia Oriental, Ásia do Sul, África do Sul, e América do Sul. Todos os clades são caracterizados por distintos polimorfismos de nucleotídeos únicos (SNPs), o que destaca o aparecimento independente e mundial deste patógeno (Lockhart et al., 2017).
Estudos comparativos em diferentes modelos animais (larvas de murino e Galleria mellonella) mostraram níveis de virulência dependentes de cepa para a C. auris. Na maioria dos casos, as cepas de C. auris provaram ser menos virulentas que as de C. albicans (Borman et al., 2016; Ben-Ami et al., 2017; Fakhim et al., 2018; Wang et al., 2018). Entretanto, os casos relatados de surtos de C. auris foram ligados a uma alta taxa de mortalidade, principalmente em pacientes imunocomprometidos. Outras condições como diabetes, doenças cardiovasculares e/ou pulmonares, sepse ou tratamento prévio com antibióticos também parecem ser fatores de risco importantes (Osei Sekyere, 2018).
Excepcionalmente para uma levedura, C. auris se espalhou com sucesso em ambientes nosocomiais. Por exemplo, num hospital do Reino Unido, um único paciente colonizado com C. auris levou a identificações subsequentes de outros casos entre outros pacientes hospitalizados. O patógeno também foi detectado na equipe de saúde mesmo vários meses após o seu primeiro isolamento, sugerindo uma transmissão eficiente de humano para humano. Além disso, foram encontrados vestígios de C. auris nas laterais do leito, nos parapeitos, monitores e outros equipamentos em todo o hospital, mostrando que a C. auris é capaz de persistir em superfícies nosocomiais (Schelenz et al., 2016), em contraste com outras espécies de Candida. Investigação de um surto em outro hospital do Reino Unido relatou que o uso de equipamentos reutilizáveis, como sondas de temperatura axilar, foi a principal causa de transmissão aos pacientes (Eyre et al., 2018), ilustrando ainda mais sua capacidade de sobreviver em superfícies.
A capacidade de sobreviver em ambientes nosocomiais poderia ser devido à capacidade de C. auris de formar biofilmes; uma forma de crescimento na qual as células são dispostas juntas em microcolônias protegidas por uma matriz de glucan. Por convenção, as células fechadas na matriz são chamadas sésseis, e as células fora dela são chamadas planctónicas. As células de C. auris protegidas por esta estrutura provaram ser difíceis de eliminar, mesmo com procedimentos de desinfecção de alto nível (Kean et al., 2018b).
Além de potencialmente permitir um aumento da sobrevivência em superfícies inertes, a capacidade de C. auris de formar biofilmes também pode ser um fator importante de sua resistência aos antifúngicos sistêmicos usados no tratamento de pacientes. De fato, o fato de células formadoras de biofilme serem isoladas de feridas e pontas de cateteres indica que algumas células de C. auris poderiam sobreviver na forma de biofilme em pacientes (Borman et al., 2016) e foi demonstrado que células sésseis apresentavam suscetibilidade reduzida para vários antifúngicos. (Sherry et al., 2017). Finalmente, o biofilme também demonstrou ser um importante fator de virulência, já que as cepas de C. auris formadoras de biofilme estão ligadas ao aumento da morbidade e mortalidade (Borman et al, 2016).
Com esta revisão objetivamos destacar mecanismos de resistência molecular adquiridos pela C. auris, o que levou à sua extraordinária sobrevida em ambiente nosocomial e em pacientes hospitalizados.
C. auris Antifungal Susceptibility
Após 10 anos de registro da concentração inibitória mínima (MIC) para antifúngicos disponíveis, o CDC publicou recentemente uma tentativa de pontos de ruptura para melhor definir a suscetibilidade à C. auris antifúngica1. Parecia claramente que a C. auris tem MICs excepcionalmente altas para as quatro principais classes de antifúngicos: azóis, equinocandinas, polenos (Figura 1B), e análogos de nucleósidos. Neste capítulo, examinaremos em detalhe dois fatores que provaram ter um papel nesta diminuição da susceptibilidade aos tratamentos antifúngicos: mecanismos de resistência molecular e mecanismos de resistência associados ao biofilme.
2.1. Mecanismos de Resistência Molecular
Definimos aqui resistência molecular como as formas pelas quais as células individuais podem diminuir sua sensibilidade aos antifúngicos devido a modificações genéticas que levam à modificação de alvos de drogas, sobreexpressão de bombas de efluxo, ou modificações do metabolismo (veja para revisão: Pemán et al., 2009; Sanglard et al., 2009; Krishnasamy et al., 2018). Esta seção compila, para cada classe de antifúngico, os principais mecanismos de resistência molecular encontrados em C. auris até o momento. Adicionalmente, a Tabela 1 resume para cada droga, o modo de ação, os mecanismos de resistência relacionados descritos em C. auris Candida e os mecanismos de resistência descobertos em C. auris, até agora.

Tabela 1. Farmacodinâmica das quatro principais classes de antifúngicos e mecanismos de resistência ligados em Candida spp. em geral e C. auris especificamente.
2.1.1. Mecanismos de resistência aos azóis
2.1.1.1. MFS e bombas de efluxo ABC sobreexpressão
Bombas de efluxo são proteínas que transportam componentes através da membrana celular. Algumas delas podem bombear drogas fora da célula, baixando sua concentração e efeito sobre a célula. Existem duas grandes famílias de bombas de efluxo envolvidas na resistência aos antifúngicos: ATP Binding Cassette (ABC) e Major Facilitator Superfamily (MFS) transportadores. A sobreexpressão aprimorada das bombas de efluxo é um dos principais mecanismos de resistência a azóis em espécies patogênicas de Candida (Schuetzer-Muehlbauer et al., 2003; Morschhäuser et al., 2007).
Cepas indianas de C. auris sequenciadas em dois estudos diferentes exibem numerosos genes ortológicos a transportadores de C. albicans ABC e MFS (Chatterjee et al., 2015; Sharma et al., 2016). Curiosamente, as cepas de C. auris que foram isoladas em Israel mostraram uma atividade intrinsecamente alta de transporte de C. glabrata e C. haemulonii, medindo efluxo de um substrato fluorescente (Rhodamine 6G) (Ben-Ami et al., 2017).
Em C. albicans, CDR1 é um gene codificador de uma bomba de efluxo de ABC, conhecido por seu papel na resistência a azole (Sanglard et al., 1995). Um gene homólogo ao CDR1 foi encontrado em C. auris. (Rybak et al., 2019). O mesmo estudo também mostrou que a deleção deste gene poderia aumentar a suscetibilidade de cepas resistentes de 64 para 128 vezes (Rybak et al., 2019).
2.1.1.2. Mutações pontuais no ERG11
Ergosterol é um componente chave da membrana em fungos. Em Candida spp., sua biossíntese é mediada, entre outros, pela enzima Lanosterol 14-alfa-dimetilase (LD), codificada pelo gene ERG11 que converte o lanosterol em ergosterol. O LD é o alvo primário dos azóis, antifúngicos que inibem a função da enzima e desligam efetivamente a biossíntese do ergosterol, prejudicando assim a integridade da membrana (Sanglard et al., 1998).
Mutações pontuais no ERG11 mostraram reduzir a sensibilidade ao azole em Candida spp, particularmente em três regiões de “ponto quente” localizadas entre os aminoácidos 105-165, 266-287, e 405-488 (Vandeputte et al., 2012).
Interessantemente, após ter recuperado as seqüências de aminoácidos de 44 C. auris isolados da Índia, 15 mutações de falta foram encontradas quando alinhadas à seqüência ERG11 do tipo selvagem de C. albicans (Chowdhary et al., 2018). Cinco destas mutações já estavam associadas à resistência azótica em C. albicans. Dentre essas variantes, duas foram encontradas em cada cepa resistente testada neste estudo, a saber Y132F ou K143R (Chowdhary et al., 2018). Pode-se também notar que estas mutações estão no primeiro “hot spot” discutido anteriormente.
Uma tela para variantes no gene ERG11 das cepas de C. auris da Columbia identificou as mesmas substituições de Y132F e K143R. Na expressão heteróloga dessas duas mutações em S. cerivisiae, MICs para azoles apresentaram um aumento de duas vezes em relação às cepas de S. cerevisiae expressando o gene ERG11 do tipo selvagem C. auris (Healey et al., 2018).
2.1.1.3. ERG11 overexpression
Em C. albicans, uma superexpressão do ERG11 também tem sido ligada a uma resistência ao tratamento com azole, 1998).
Em C. auris, experimentos PCR em tempo real mostraram que, na ausência de fluconazol, não houve diferença na expressão do ERG11 entre cepas sensíveis ao fluconazol e cepas resistentes ao fluconazol (Chowdhary et al., 2018). Na presença do antifúngico, a expressão do ERG11 aumenta em cepas resistentes quando comparada a um controle sem fluconazol (Chowdhary et al., 2018). Entretanto, o efeito deste medicamento na expressão do ERG11 não foi testado em cepas suscetíveis. Portanto, a expressão condicionalmente aumentada do ERG11 na presença de fluconazol não demonstrou ser específica para cepas resistentes, até o momento.
2.1.2. Mecanismos de Resistência às Equinocandinas
Beta(1,3)D-glucan é um componente chave da parede celular fúngica e é codificado pelos genes FKS1 e FKS2, duas subunidades da Beta(1,3)D-glucan sintetase. As equinocandinas inibem esta enzima, diminuindo a quantidade de glucanos na parede celular (Martins et al., 2011).
Em C. albicans e outras espécies não auris Candida, várias mutações que levam à resistência à equinocandina foram encontradas nas mesmas duas regiões de FKS1 e FKS2. Assim, foram denominadas “hot-spots” 1 e 2 (HS1 e HS2). No gene C. albicans FKS1 estes “hot-spots” estão localizados entre os aminoácidos 641-649 e 1.345-1.365 (Park et al., 2005). A sequência das regiões dos pontos quentes correspondentes das cepas de 38 C. auris leva à descoberta de uma substituição de aminoácidos S639F que está correlacionada com a resistência à pan-echinocandina: ela estava presente em todas as quatro cepas resistentes e ausente nas 34 susceptíveis. Esta posição em C. auris FKS1 está alinhada com a posição 645 (também uma Serine) em C. albicans (Chowdhary et al., 2018). Curiosamente, esta mutação está na região alinhada com o HS1 de C. albicans FKS1. Outros estudos observaram diferentes mutações no mesmo local em cepas de C. auris resistentes à equinocandina: S639Y e S639P (Rhodes et al., 2018). Esta última foi confirmada para causar resistência à equinocandina in vivo em um modelo de rato (Kordalewska et al., 2018).
FKS2 também foi encontrada em uma única cópia no genoma de C. auris (Sharma et al., 2016), mas nenhuma mutação associada à resistência à equinocandina foi encontrada neste gene para nosso conhecimento.
2.1.3. Mecanismos de Resistência aos Polígenos
Como mostrado na Figura 1B, várias cepas de C. auris são resistentes à Anfotericina B (AMB). Na espécie Candida, modificações da composição do esterol da membrana foram destacadas como mecanismo de resistência (Haynes et al., 1996; Nolte et al., 1997). Em C. albicans, mutações no ERG 2, 3, 5, 6 ou 11 mostraram ter esse efeito (Arendrup e Patterson, 2017). Rhodes et al. triado 27 C. auris isolados do Reino Unido para SNPs nestes genes em cepas que apresentam sensibilidade reduzida à AMB. Entretanto, não foram encontradas variantes explicando essas diferenças na suscetibilidade a drogas (Rhodes et al., 2018).
2.1.4. Mecanismos de Resistência à Flucitosina (5-Fluorocitosina)
Flucitosina é um análogo nucleósido que inibe a síntese de ácido nucléico. Após a entrada da célula, a flucitosina tem de ser activada para ter um efeito antifúngico. Esta ativação requer, entre outras, a proteína codificada pelo gene FUR1 Waldorf e Polak (1983). Em espécies não auris Candida mutações em FUR1 foram demonstradas como ligadas à resistência à flucitosina (Vandeputte et al., 2012).
Rhodes et al. sequenciaram uma cepa de C. auris resistente à flucitosina e observaram uma substituição de aminoácidos F211I no gene FUR1 (Rhodes et al., 2018). Esta mutação específica falsa não tem equivalente conhecido em outras espécies de Candida, portanto estudos adicionais são necessários para determinar se esta mutação é a causa da resistência à flucitosina na cepa testada de C. auris (Rhodes et al., 2018). Entretanto, este medicamento é menos empregado que outros antifúngicos. Por causa disso, menos estudos foram realizados para entender melhor a resistência de C. auris a este composto.
Para resumir, alguns mecanismos de resistência previamente descobertos em espécies de Candida como C. glabrata e C. albicans, também foram mostrados por C. auris. Eles estão listados na última coluna da Tabela 1. Entretanto, a resistência a C. auris antifúngica é apenas parcialmente explicada por estes mecanismos de resistência previamente descritos. A fim de eventualmente projetar novos agentes terapêuticos e, de modo mais geral, melhorar o cuidado ao paciente, são necessários mais estudos para entender melhor os mecanismos subjacentes que levam às gamas excepcionalmente altas de C. auris apresentadas por alguns C. auris (ver Figura 1B).
2.2. Biofilme: Uma Forma de Vida Resistente a Antifúngicos
Foi mostrado que os MICs para vários antifúngicos são mais altos em células sésseis de C. auris (até 4, 20, 60 vezes para voriconazol, anfotericina B, e Micafungin, respectivamente) em comparação com células planctônicas (Sherry et al., 2017). Da mesma forma, as concentrações mínimas de erradicação do biofilme mostraram ser 512 vezes maiores do que as MIC para equinocandinas e azoles (Romera et al., 2019). Este fenômeno foi mostrado anteriormente para C. albicans (Hawser e Douglas, 1995). Os mecanismos moleculares responsáveis por esses MICs elevados ainda são largamente desconhecidos em C. auris, mas alguns estudos já forneceram alguns insights.
MSF e ABC genes codificadores de efluxo foram encontrados upregulados (2 a 4 vezes) em células sésseis comparadas com células planctônicas (Kean et al., 2018a). Concordantemente, a atividade das proteínas correspondentes foi aumentada em duas vezes. Entretanto, quando tratadas com inibidores da bomba de efluxo a sensibilidade antifúngica das células sésseis aumentou 4 a 16 vezes após 12 h. Isto sugere que as bombas de efluxo desempenham um papel importante na resistência aos antifúngicos exibida por este tipo de células (Kean et al., 2018a).
Em C. albicans, a matriz do exopolímero é conhecida por se ligar não especificamente a todas as classes de antifúngicos e sequestrá-los fora das células (Nett et al., 2010). Como as Candida spp. compartilham um perfil de polissacarídeo do núcleo, foi sugerido que este mecanismo poderia ser conservado também em C. auris (Kean et al., 2018a). Isto foi posteriormente confirmado em ensaios in vivo e in vitro (Dominguez et al., 2019).
PersistênciaNosocomial
A capacidade da C. auris de colonizar e persistir em superfícies é excepcional: ela é capaz de persistir mais tempo em superfícies úmidas em comparação com C. albicans (Piedrahita et al., 2017). Ela também tem uma atividade metabólica prolongada em superfícies, semelhante à C. parapsilosis, um conhecido colonizador de pele e plástico (Piedrahita et al., 2017; Welsh et al., 2017).
O aumento da sobrevivência de C. auris em superfícies foi investigado por dois estudos. O primeiro alegou que poderia estar ligado a uma maior resistência ao stress ambiental à temperatura e a outros factores de stress (Kean et al., 2018b). O segundo pressupõe que isso poderia ser devido à formação do biofilme. No entanto, isso ainda não foi comprovado, pois, até agora, nenhum biofilme isolado foi recuperado das superfícies ambientais (Ku et al., 2018). Deve-se notar que estas duas possibilidades não são mutuamente exclusivas, e que a formação do biofilme pode até ser a causa do aumento da resistência ao estresse (Ku et al., 2018).
A persistência da C. auris em ambientes nosocomiais é aumentada pela resistência deste patógeno aos procedimentos de desinfecção. A eficácia do NaOCl e do ácido peracético em superfícies de aço inoxidável, polímero (lamelas de poliéster) e celulose foi testada. Ambos os desinfectantes mostraram uma eficácia significativa para matar as células de C. auris em todas as superfícies. No entanto, algumas células viáveis permaneceram após a aplicação de NaOCl em superfícies não porosas (lamelas de aço inoxidável e de poliéster). De facto, foram necessárias concentrações mais elevadas deste desinfectante associadas a tempos de exposição mais longos para diminuir o recrescimento, mas mesmo assim, não foram capazes de erradicar completamente o patogéneo. Por exemplo, uma concentração de 10.000 partes por milhão (ppm) de NaOCl acoplada a uma exposição de 5 minutos em aço inoxidável, foi suficiente para observar uma redução significativa das colônias. Entretanto, observou-se um recrescimento substancial após a reinoculação em um meio rico (Kean et al., 2018b).
Para o ácido peracético, que é um desinfetante de alto nível, esse recrescimento foi dependente da superfície, já que não se observou recrescimento para o polímero em oposição ao aço inoxidável. Eles realizaram as mesmas experiências com C. glabrata e C. albicans e obtiveram resultados semelhantes (Kean et al., 2018b).
Isto mostra que os procedimentos de desinfecção de alto nível podem ser insuficientes para uma limpeza completa da superfície e que as orientações pré-estabelecidas para a desinfecção da superfície têm de ser adaptadas a estas espécies de Candida (Kean et al., 2018b). Fatores como o tipo de superfícies e tempos de exposição devem ser considerados ao implementar estes procedimentos de desinfecção (Kean et al., 2018b; Ku et al., 2018) e não apenas o custo e a facilidade de uso, como é freqüentemente o caso (Ku et al., 2018).
Baseado nos resultados de dois estudos (Cadnum et al., 2017; Rutala et al., 2019), o CDC recomenda o uso de desinfetantes eficazes contra esporos de Clostridioides difficile. Se não for possível, sugerem alternativas como peróxido de hidrogênio 0,5-1,4% ou compostos de amônio quaternário suplementados com álcool isopropílico e/ou álcool etílico2. Finalmente, a luz ultravioleta C também pode ser um candidato interessante para a desinfecção de superfícies. Ela provou ser altamente eficaz para matar colônias de C. auris, dado o tempo de exposição e distância suficientes. Entretanto, estudos adicionais seriam necessários antes de implementar tais métodos em ambientes hospitalares (de Groot et al., 2019).
Conclusão
As capacidades patogênicas e virulentas da C. auris são profundamente preocupantes. Especialmente porque os surtos ocorreram independentemente em diferentes regiões do mundo e que os isolados associados mostraram menor susceptibilidade aos antifúngicos mais comumente usados no tratamento de pacientes (ver Figura 1).
Para explicar a reduzida susceptibilidade aos antifúngicos em C. auris, pesquisadores fizeram uso do conhecimento ampliado já disponível sobre outras espécies de C. auris, particularmente C. albicans. Como resultado, alguns dos mecanismos de resistência de C. auris foram destacados (ver Tabela 1). Mais precisamente, ficou provado que duas mutações pontuais no ERG11 e a superexpressão do transportador ABC Cdr1 diminuíram a sensibilidade ao fluconazol. Além disso, uma substituição de aminoácidos no FKS1 demonstrou reduzir a sensibilidade da C. auris às equinocandinas, que normalmente é a droga de escolha para o tratamento contra este patógeno (Bidaud et al., 2018). Finalmente, uma mutação em FUR1 foi identificada em uma cepa de C. auris resistente à flucytosina, mas ainda não foi demonstrada como a causa desta sensibilidade reduzida. Pensa-se que o biofilme é um mecanismo de resistência per se, pois demonstrou diminuir a susceptibilidade às drogas. No entanto, ainda são necessárias mais pesquisas para descrever toda a variedade de processos que ocorrem nesta forma de crescimento. É importante notar que o biofilme é uma estrutura complexa que permanece mal compreendida e difícil de investigar, mesmo em espécies bem conhecidas como a C. albicans.
As propriedades de resistência da C. auris estão associadas a uma propensão para causar infecções nosocomiais. A sua capacidade de aderir a uma variedade de superfícies nosocomiais e sobreviver à desinfecção são factores críticos de propagação. A presença de múltiplos tipos de superfícies representa um desafio para as instituições clínicas, pois os procedimentos de desinfecção padrão são ineficientes e desadaptados para cada tipo de superfície. Este problema, também surge da pesquisa insuficiente sobre este assunto. Além disso, um melhor entendimento dos mecanismos subjacentes à resistência desinfetante da C. auris ajudaria a melhorar os protocolos de higiene e evitar futuros surtos nosocomiais.
Contribuições dos autores
FC, AG, e LJ participaram igualmente da redação desta revisão. AC supervisionou e participou da redação desta revisão.
Conflito de Interesses
Os autores declaram que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Agradecimentos
Gostaríamos de agradecer a Clara Heiman como falante nativa de inglês por ter revisado nosso manuscrito.
Notas de rodapé
1. ^CDC (2019). Antifungal Susceptibility Testing and Interpretation (Teste de Susceptibilidade Antifúngica e Interpretação). Disponível online em: https://www.cdc.gov/fungal/candida-auris/c-auris-antifungal.html (acedido a 29 de Abril de 2019).
2. ^CDC (2018). Prevenção e Controle de Infecções para Candida auris. Disponível online em: https://www.cdc.gov/fungal/candida-auris/c-auris-infection-control.html (acedido a 12 de Novembro de 2019).
Abdalhamid, B., Almaghrabi, R., Althawadi, S., e Omrani, A. (2018). Primeiro relato de infecções por Candida auris da Arábia Saudita. J. Infect. Public Health 11, 598-599. doi: 10.1016/j.jiph.2018.05.010
PubMed Abstract | CrossRef Full Text | Google Scholar
Adam, R. D., Revathi, G., Okinda, N., Fontaine, M., Shah, J., Kagotho, E., et al. (2019). Análise da fungemia de Candida auris em uma única instalação no Quênia. Int. J. Infect. Dis. 85, 182-187. doi: 10.1016/j.ijid.2019.06.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Adams, E., Quinn, M., Tsay, S., Poirot, E., Chaturvedi, S., Southwick, K., et al. (2018). Candida auris em estabelecimentos de saúde, Nova Iorque, EUA, 2013-2017. Emergente. Infecto. Dis. 24, 1816-1824. doi: 10.3201/eid2410.180649
CrossRef Full Text | Google Scholar
Arendrup, M. C., e Patterson, T. F. (2017). Candida multirresistente: epidemiologia, mecanismos moleculares, e tratamento. J. Infect. Dis. 216, S445-S451. doi: 10.1093/infdis/jix131
PubMed Abstract | CrossRef Full Text | Google Scholar
Barantsevich, N. E., Orlova, O. E., Shlyakhto, E. V., Johnson, E. M., Woodford, N., Lass-Floerl, C., et al. (2019). Emergência de Candida auris na Rússia. J. Hospital. Infect. 102, 445-448. doi: 10.1016/j.jhin.2019.02.021
PubMed Abstract | CrossRef Full Text | Google Scholar
Belkin, A., Gazit, Z., Keller, N., Ben-Ami, R., Wieder-Finesod, A., Novikov, A., et al. (2018). Candida auris infection leading to nosocomial transmission, Israel, 2017. Emergente. Infectada. Dis. 24, 801-804. doi: 10.3201/eid2404.171715
PubMed Abstract | CrossRef Full Text | Google Scholar
Ben-Ami, R., Berman, J., Novikov, A., Bash, E., Shachor-Meyouhas, Y., Zakin, S., et al. (2017). Multidrug-resistente Candida haemulonii e C. auris, Tel Aviv, Israel. Emergentes. Infectados. Dis. 23, 195-203. doi: 10.3201/eid2302.161486
PubMed Abstract | CrossRef Full Text | Google Scholar
Bidaud, A. L., Chowdhary, A., e Dannaoui, E. (2018). Candida auris: uma levedura emergente resistente a drogas – Uma mini-revisão. J. Mycol. Méd. 28, 568-573. doi: 10.1016/j.mycmed.2018.06.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Biswal, M., Rudramurthy, S. M., Jain, N., Shamanth, A. S., Sharma, D., Jain, K., et al. (2017). Controlando um possível surto de infecção por Candida auris: lições aprendidas de múltiplas intervenções. J. Hosp. Infect. 97, 363-370. doi: 10.1016/j.jhin.2017.09.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Borman, A. M., Szekely, A., and Johnson, E. M. (2016). Patogenicidade comparativa dos isolados do Reino Unido do patógeno emergente Candida auris e outras espécies patogênicas chave. mSphere 1:e00189-16. doi: 10.1128/mSphere.00189-16
CrossRef Full Text | Google Scholar
Cadnum, J. L., Shaikh, A. A., Piedrahita, C. T., Sankar, T., Jencson, A. L., Larkin, E. L., et al. (2017). Eficácia dos desinfetantes contra Candida auris e outras espécies de candida. Infect. Control Hosp. Epidemiol. 38, 1240-1243. doi: 10.1017/ice.2017.162
PubMed Abstract | CrossRef Full Text | Google Scholar
Chatterjee, S., Alampalli, S. V., Nageshan, R. K., Chettiar, S. T., Joshi, S., and Tatu, U. S. (2015). Genoma rascunho de um patógeno multirresistente Candida auris, comumente mal diagnosticado. BMC Genomics 16:686. doi: 10.1186/s12864-015-1863-z
PubMed Abstract | CrossRef Full Text | Google Scholar
Chow, N. A., Gade, L., Tsay, S. V., Forsberg, K., Greenko, J. A., Southwick, K. L., et al. (2018). Múltiplas introduções e subsequente transmissão de Candida auris multirresistente nos EUA: um levantamento epidemiológico molecular. Lancet Infect. Dis. 18, 1377-1384. doi: 10.1016/S1473-3099(18)30597-8
PubMed Abstract | CrossRef Full Text | Google Scholar
Chowdhary, A., Prakash, A., Sharma, C., Kordalewska, M., Kumar, A., Sarma, S., et al. (2018). Um estudo multicêntrico dos padrões de susceptibilidade a antifúngicos entre 350 isolados de Candida auris (2009-17) na Índia: papel dos genes ERG11 e FKS1 na resistência à azole e equinocandina. J. Antimicrob. Quimioterapia. 73, 891-899. doi: 10.1093/jac/dkx480
PubMed Abstract | CrossRef Full Text | Google Scholar
de Groot, T., Chowdhary, A., Meis, J. F., e Voss, A. (2019). Killing of Candida auris by UV-C: importance of exposure time and distance. Mycoses 62, 408-412. doi: 10.1111/myc.12903
PubMed Abstract | CrossRef Full Text | Google Scholar
Desoubeaux, G., Bailly, E., Guillaume, C., De Kyvon, M.-A., Tellier, A.-C., Morange, V., et al. (2018). Candida auris nos laboratórios de micologia contemporânea: alguns truques práticos para identificá-la de forma confiável de acordo com uma experiência francesa recente. J. Mycol. Med. 28, 407-410. doi: 10.1016/j.mycmed.2018.02.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Dominguez, E. G., Zarnowski, R., Choy, H. L., Zhao, M., Sanchez, H., Nett, J. E., et al. (2019). Papel conservado para polissacarídeos de matriz de biofilme na resistência a drogas Candida auris. mSphere 4:e00680-18. doi: 10.1128/mSphereDirect.00680-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Escandón, P., Chow, N. A., Caceres, D. H., Gade, L., Berkow, E. L., Armstrong, P., et al. (2019). A epidemiologia molecular de Candida auris na colômbia revela uma colonização altamente relacionada, em todo o país, com padrões regionais de resistência à anfotericina B. Clin. Infect. Dis. 68, 15-21. doi: 10.1093/cid/ciy411
CrossRef Full Text | Google Scholar
Eyre, D. W., Sheppard, A. E., Madder, H., Moir, I., Moroney, R., Quan, T. P., et al. (2018). Um surto de Candida auris e seu controle em um ambiente de cuidados intensivos. N. Engl. J. Med. 379, 1322-1331. doi: 10.1056/NEJMoa1714373
PubMed Abstract | CrossRef Full Text | Google Scholar
Fakhim, H., Vaezi, A., Dannaoui, E., Chowdhary, A., Nasiry, D., Faeli, L., et al. (2018). Virulência comparativa de Candida auris com Candida haemulonii, Candida glabrata e Candida albicans em um modelo murino. Mycoses 61, 377-382. doi: 10.1111/myc.12754
PubMed Abstract | CrossRef Full Text | Google Scholar
Hawser, S. P., e Douglas, L. J. (1995). Resistência dos biofilmes de Candida albicans aos agentes antifúngicos in vitro. Antimicrob. Agentes Quimioterápicos. 39, 2128-2131. doi: 10.1128/AAC.39.9.2128
PubMed Abstract | CrossRef Full Text | Google Scholar
Haynes, M. P., Chong, P. L.-G., Buckley, H. R., e Pieringer, R. A. (1996). Estudos de fluorescência sobre a ação molecular da anfotericina B em células fúngicas suscetíveis e resistentes. Biochemistry 35, 7983-7992. doi: 10.1021/bi952910c
PubMed Abstract | CrossRef Full Text | Google Scholar
Healey, K. R., Kordalewska, M., Ortigosa, C. J., Singh, A., Berrío, I., Chowdhary, A., et al. (2018). As mutações ERG11 limitadas identificadas em isolados de Candida auris contribuem directamente para a redução da susceptibilidade azole. Antimicrob. Agentes Quimioterápicos. 62, e01427-18. doi: 10.1128/AAC.01427-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Jackson, B. R., Chow, N., Forsberg, K., Litvintseva, A. P., Lockhart, S. R., Welsh, R., et al. (2019). Sobre as origens de uma espécie: o que poderia explicar a ascensão de Candida auris? J. Fungi 5:E58. doi: 10.3390/jof5030058
PubMed Abstract | CrossRef Full Text | Google Scholar
Kean, R., Delaney, C., Sherry, L., Borman, A., Johnson, E. M., Richardson, M. D., et al. (2018a). Transcriptome assembly and profiling of Candida auris reveals novel insights into biofilm-mediated resistance. mSphere 3:e00334-18. doi: 10.1128/mSphere.00334-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Kean, R., Sherry, L., Townsend, E., McKloud, E., Short, B., Akinbobola, A., et al. (2018b). Desafios de desinfecção superficial para Candida auris: um estudo in-vitro. J. Hospital. Infectar. 98, 433-436. doi: 10.1016/j.jhin.2017.11.015
PubMed Abstract | CrossRef Full Text | Google Scholar
Kenters, N., Kiernan, M., Chowdhary, A., Denning, D. W., Pemán, J., Saris, K., et al. (2019). Controle de Candida auris em instituições de saúde. Resultado de uma reunião de peritos do ISAC. Int. J. Antimicrob. Agentes 54, 400-406. doi: 10.1016/j.ijantimicag.2019.08.013
CrossRef Full Text | Google Scholar
Kordalewska, M., Lee, A., Park, S., Berrio, I., Chowdhary, A., Zhao, Y., et al. (2018). Entendendo a resistência à equinocandina no patógeno emergente Candida auris. Antimicrob. Agentes quimioterápicos. 62:e00238-18. doi: 10.1128/AAC.00238-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Krishnasamy, L., Krishnakumar, S., Kumaramanickavel, G., e Saikumar, C. (2018). Mecanismos moleculares de resistência a drogas antifúngicas em espécies de Candida. J. Clin. Diagnóstico. Res. 12, DE01-DE06. doi: 10.7860/JCDR/2018/36218.11961
CrossRef Full Text | Google Scholar
Ku, T. S. N., Walraven, C. J., e Lee, S. A. (2018). Candida auris: desinfectantes e implicações para o controlo de infecções. Frente. Microbiol. 9:726. doi: 10.3389/fmicb.2018.00726
CrossRef Full Text | Google Scholar
Leach, L., Russell, A., Zhu, Y., Chaturvedi, S., e Chaturvedi, V. (2019). Um ensaio rápido e automatizado de PCR em tempo real Candida auris para testes de alto rendimento de amostras de vigilância com sistema aberto BD MAX™. J. Clin. Microbiol. 57:e00630-19. doi: 10.1128/JCM.00630-19
CrossRef Full Text | Google Scholar
Lesho, E. P., Bronstein, M. Z., McGann, P., Stam, J., Kwak, Y., Maybank, R., et al. (2018). Importação, mitigação e epidemiologia genômica de Candida auris em um grande hospital universitário. Infectar. Epidemiol do hospital de controle. 39, 53-57. doi: 10.1017/ice.2017.231
PubMed Abstract | CrossRef Full Text | Google Scholar
Lockhart, S. R., Etienne, K. A., Vallabhaneni, S., Farooqi, J., Chowdhary, A., Govender, N. P., et al. (2017). Emergência simultânea de Candida auris multirresistente em 3 continentes confirmada por seqüenciamento de genomas inteiros e análises epidemiológicas. Clin. Infectar. Dis. 64, 134-140. doi: 10.1093/cid/ciw691
PubMed Abstract | CrossRef Full Text | Google Scholar
Lopez-Ribot, J. L., McAtee, R. K., Lee, L. N., Kirkpatrick, W. R., White, T. C., Sanglard, D., et al. (1998). Distintos padrões de expressão gênica associados ao desenvolvimento de resistência ao fluconazol em Candida albicans em série isolados de pacientes infectados pelo vírus da imunodeficiência humana com candidíase orofaríngea. Antimicrob. Agentes Quimioterápicos. 42, 2932-2937. doi: 10.1128/AAC.42.11.2932
PubMed Abstract | CrossRef Full Text | Google Scholar
Martins, I. M., Cortés, J. C. G., Muñoz, J., Moreno, M. B., Ramos, M., Clemente-Ramos, J. A., et al. (2011). Atividades diferenciais de três famílias de inibidores específicos de beta(1,3)glucan sintase em cepas de levedura de fissão do tipo selvagem e resistentes. J. Biol. Chem. 286, 3484-3496. doi: 10.1074/jbc.M110.174300
PubMed Abstract | CrossRef Full Text | Google Scholar
Mayor, S. (2018). O surto de Candida auris foi ligado a sondas de temperatura axilar reutilizáveis, achados do estudo. BMJ 363:k4133. doi: 10.1136/bmj.k4133
PubMed Abstract | CrossRef Full Text | Google Scholar
Morschhäuser, J., Barker, K. S., Liu, T. T., Bla-Warmuth, J., Homayouni, R., and Rogers, P. D. (2007). O fator de transcrição Mrr1p controla a expressão da bomba de efluxo MDR1 e mede a resistência a múltiplas drogas em Candida albicans. PLoS Pathog. 3:e164. doi: 10.1371/journal.ppat.0030164
PubMed Abstract | CrossRef Full Text | Google Scholar
Nett, J. E., Crawford, K., Marchillo, K., and Andes, D. R. (2010). Papel do Fks1p e do glucano de matriz em Candida albicans biofilme de resistência à equinocandina, pirimidina e polietileno. Antimicrob. Agentes quimioterápicos. 54, 3505-3508. doi: 10.1128/AAC.00227-10
PubMed Abstract | CrossRef Full Text | Google Scholar
Nolte, F. S., Parkinson, T., Falconer, D. J., Dix, S., Williams, J., Gilmore, C., et al. (1997). Isolamento e caracterização de fluconazol e anfotericina B resistente à Candida albicans a partir do sangue de dois pacientes com leucemia. Antimicrob. Agentes Quimioterápicos. 41, 196-199. doi: 10.1128/AAC.41.1.196
PubMed Abstract | CrossRef Full Text | Google Scholar
O’Connor, C., Bicanic, T., Dave, J., Evans, T. J., Moxey, P., Adamu, U., et al. (2019). Erupção de Candida auris numa ala vascular – a chegada inesperada de um patogénico previsto. J. Hospital. Infectar. 103, 106-108. doi: 10.1016/j.jhin.2019.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Osei Sekyere, J. (2018). Candida auris: uma revisão sistemática e meta-análise de atualizações atuais sobre um patógeno multirresistente emergente. MicrobiologyOpen 7:e00578. doi: 10.1002/mbo3.578
PubMed Abstract | CrossRef Full Text | Google Scholar
Park, J. Y., Bradley, N., Brooks, S., Burney, S., and Wassner, C. (2019). Gerenciamento de pacientes com Candida auris fungemia no hospital comunitário, Brooklyn, Nova Iorque, EUA, 2016-20181. Emergente. Infecto. Dis. 25, 601-602. doi: 10.3201/eid2503.180927
PubMed Abstract | CrossRef Full Text | Google Scholar
Park, S., Kelly, R., Kahn, J. N., Robles, J., Hsu, M.-J., Register, E., et al. (2005). As substituições específicas no alvo Fks1p da equinocandina são responsáveis pela redução da susceptibilidade dos isolados de Candida sp. clínicos e laboratoriais raros. Antimicrob. Agentes quimioterápicos. 49, 3264-3273. doi: 10.1128/AAC.49.8.3264-3273.2005
CrossRef Full Text | Google Scholar
Parra-Giraldo, C. M., Valderrama, S. L., Cortes-Fraile, G., Garzón, J. R., Ariza, B. E., Morio, F., et al. (2018). Primeiro relato de casos esporádicos de Candida auris na Colômbia. Int. J. Infect. Dis. 69, 63-67. doi: 10.1016/j.ijid.2018.01.034
PubMed Abstract | CrossRef Full Text | Google Scholar
Pemán, J., Cantón, E., e Espinel-Ingroff, A. (2009). Mecanismos de resistência aos medicamentos antifúngicos. Especialista Rev. Antifúngico. Ter. 7, 453-460. doi: 10.1586/eri.09.18
PubMed Abstract | CrossRef Full Text | Google Scholar
Piedrahita, C. T., Cadnum, J. L., Jencson, A. L., Shaikh, A. A. A., Ghannoum, M. A., e Donskey, C. J. (2017). As superfícies ambientais nas instalações de saúde são uma fonte potencial para a transmissão de Candida auris e outras espécies de candida. Infect. Epidemiol do hospital de controle. 38, 1107-1109. doi: 10.1017/ice.2017.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Rhodes, J., Abdolrasouli, A., Farrer, R. A., Cuomo, C. A., Aanensen, D. M., Armstrong-James, D., et al. (2018). Epidemiologia genómica do surto de Candida auris, o patogéneo humano emergente. Emergente. Infecção por micróbios. 7:43. doi: 10.1101/201343
CrossRef Full Text | Google Scholar
Romera, D., Aguilera-Correa, J. J., Gadea, I., Viñuela-Sandoval, L., García-Rodríguez, J., e Esteban, J. (2019). Candida auris: uma comparação entre a susceptibilidade do planctónico e do biofilme aos antifúngicos. J. Med. Microbiol. 68, 1353-1358. doi: 10.1099/jmm.0.001036
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A., Martínez, H., Moret, A. M., Calabuig, E., Tasias, M., Alastruey-Izquierdo, A., et al. (2019a). Detecção e tratamento de Candida auris em situação de surto: fatores de risco para desenvolver colonização e candidemia por esta nova espécie em pacientes criticamente enfermos. Especialista Rev. Anti-infeccioso. Ter. 17, 295-305. doi: 10.1080/14787210.2019.1592675
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A., Moret, A. M., Tasias-Pitarch, M., Aleixandre-López, A. I., Martínez-Morel, H., Calabuig, E., et al. (2018). Um surto devido a Candida auris com colonização prolongada e candidaemia em um hospital europeu de cuidados terciários. Mycoses 61, 498-505. doi: 10.1111/myc.12781
PubMed Abstract | CrossRef Full Text | Google Scholar
Ruiz-Gaitán, A. C., Cantón, E., Fernández-Rivero, M. E., Ramírez, P., e Pemán, J. (2019b). Surto de Candida auris na Espanha: uma comparação da atividade antifúngica por três métodos com dados publicados. Int. J. Antimicrob. Agentes 53, 541-546. doi: 10.1016/j.ijantimicag.2019.02.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Rutala, W. A., Kanamori, H., Gergen, M. F., Sickbert-Bennett, E. E., e Weber, D. J. (2019). Susceptibilidade de Candida auris e Candida albicans a 21 germicidas usados em estabelecimentos de saúde. Infect. Epidemiol do hospital de controle. 40, 380-382. doi: 10.1017/ice.2019.1
PubMed Abstract | CrossRef Full Text | Google Scholar
Rybak, J. M., Doorley, L. A., Nishimoto, A. T., Barker, K. S., Palmer, G. E., e Rogers, P. D. (2019). Abrogation of triazole resistance upon deletion of CDR1 in a clinical isolate of Candida auris. Antimicrob. Agentes Quimioterápicos. 63, e00057-19. doi: 10.1128/AAC.00057-19
PubMed Abstract | CrossRef Full Text | Google Scholar
Sana, F., Hussain, W., Zaman, G., Satti, L., Khurshid, U., e Khadim, M. T. (2019). Relatório de surto de Candida auris do Paquistão: uma história de sucesso no controle de infecções em UTIs de um hospital de cuidados terciários. J. Hosp. Infect. 103, 108-110. doi: 10.1016/j.jhin.2019.06.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Sanglard, D., Coste, A., e Ferrari, S. (2009). Mecanismos de resistência a drogas antifúngicas em patógenos fúngicos a partir da perspectiva da regulação gênica transcripcional. FEMS Yeast Res. 9, 1029-1050. doi: 10.1111/j.1567-1364.2009.00578.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Sanglard, D., Ischer, F., Koymans, L., and Bille, J. (1998). Substituições de aminoácidos no citocromo P-450 lanosterol 14-demetilase (CYP51a1) de isolados clínicos de Candida albicans resistentes a azole contribuem para a resistência a agentes antifúngicos azólicos. Antimicrob. Agentes quimioterápicos. 42, 241-253.
Google Scholar
Sanglard, D., Kuchler, K., Ischer, F., Pagani, J. L., Monod, M., e Bille, J. (1995). Mecanismos de resistência aos agentes antifúngicos azólicos em isolados de Candida albicans de doentes com SIDA envolvem transportadores multi-drogas específicos. Antimicrob. Agentes Quimioterápicos. 39, 2378-2386. doi: 10.1128/AAC.39.11.2378
PubMed Abstract | CrossRef Full Text | Google Scholar
Sayeed, M. A., Farooqi, J., Jabeen, K., Awan, S., and Mahmood, S. F. (2019). Espectro clínico e fatores de impacto dos resultados de Candida auris: um único estudo central do Paquistão. BMC Infect. Dis. 19:384. doi: 10.1186/s12879-019-3999-y
PubMed Abstract | CrossRef Full Text | Google Scholar
Schelenz, S., Hagen, F., Rhodes, J. L., Abdolrasouli, A., Chowdhary, A., Hall, A., et al. (2016). Primeiro surto hospitalar da emergente Candida auris global em um hospital europeu. Antimicrob. Resistir. Infect. Control 5:35. doi: 10.1186/s13756-016-0132-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Schuetzer-Muehlbauer, M., Willinger, B., Egner, R., Ecker, G., and Kuchler, K. (2003). Reversão da resistência antifúngica mediada por bombas de efluxo ABC de Candida albicans funcionalmente expressas em levedura. Int. J. Antimicrob. Agentes 22, 291-300. doi: 10.1016/S0924-8579(03)00213-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Sexton, D. J., Kordalewska, M., Bentz, M. L., Welsh, R. M., Perlin, D. S., e Litvintseva, A. P. (2018). Detecção direta do patógeno fúngico emergente Candida auris em esfregaços clínicos de pele através do ensaio de PCR quantitativa SYBR de base verde. J. Clin. Microbiol. 56:e01337-18. doi: 10.1128/JCM.01337-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sharma, C., Kumar, N., Pandey, R., Meis, J. F., e Chowdhary, A. (2016). O sequenciamento do genoma inteiro de isolados de Candida auris multirresistentes emergentes na Índia demonstra baixa variação genética. Novos micróbios Novo Infecto. 13, 77-82. doi: 10.1016/j.nmni.2016.07.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Sherry, L., Ramage, G., Kean, R., Borman, A., Johnson, E. M., Richardson, M. D., et al. (2017). Capacidade de formação de biofilme de Candida auris altamente virulenta e multirresistente. Emergente. Infectada. Dis. 23, 328-331. doi: 10.3201/eid2302.161320
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandeputte, P., Ferrari, S., e Coste, A. T. (2012). Resistência antifúngica e novas estratégias para controlar as infecções fúngicas. Int. J. Microbiol. 2012:713687. doi: 10.1155/2012/713687
PubMed Abstract | CrossRef Full Text | Google Scholar
Waldorf, A. R., e Polak, A. (1983). Mecanismos de ação da 5-fluorocitosina. Antimicrob. Agentes quimioterápicos. 23, 79-85. doi: 10.1128/AAC.23.1.79
PubMed Abstract | CrossRef Full Text | Google Scholar
Wang, X., Bing, J., Zheng, Q., Zhang, F., Liu, J., Yue, H., et al. (2018). O primeiro isolado de Candida auris na China: aspectos clínicos e biológicos. Microbios Emergentes & Infecções 7, 1-9. doi: 10.1038/s41426-018-0095-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Welsh, R. M., Bentz, M. L., Shams, A., Houston, H., Lyons, A., Rose, L. J., et al. (2017). Sobrevivência, persistência e isolamento da levedura patogênica multirresistente emergente Candida auris em uma superfície plástica de cuidados de saúde. J. Clin. Microbiol. 55, 2996-3005. doi: 10.1128/JCM.00921-17
PubMed Abstract | CrossRef Full Text | Google Scholar
Leave a Reply