Frontiers in Celland Developmental Biology
Background
A unidade básica do aparelho do Golgi é geralmente considerada como uma pilha de cisternas que é altamente polarizada, com o cis-face recebendo a carga das ER e a trans-face da pilha, o TGN, associado à classificação de carga para exportação pós-Golgi (Boncompain e Perez, 2013). No entanto, a organização destas mini-pilhetas na célula varia entre os diferentes organismos. Nas plantas e invertebrados, as pilhas individuais de Golgi estão espalhadas independentemente pelo citoplasma, enquanto na maioria das células vertebradas durante a interfase, as pilhas individuais de Golgi são fundidas em uma estrutura compacta de fitas localizada nas proximidades do MTOC (Wei e Seemann, 2017). A estrutura da fita de Golgi em células de mamíferos é melhor revelada por microscopia eletrônica (Rambourg e Clermont, 1997); a microscopia óptica de alta resolução usando marcadores cis e trans também pode detectar a organização da fita (Gosavi et al., 2018). Uma questão importante em investigação no campo é a relevância da estrutura da fita Golgi e as diferenças funcionais da fita Golgi em comparação com as “mini-fitas Golgi isoladas” ou outros estados da arquitetura Golgi. Em outras palavras, qual é a vantagem evolutiva da morfologia mais complexa das fitas do aparelho de Golgi em células vertebradas e quais funções podem ser reguladas por uma transição para uma estrutura não ribbon?
Estudos nos últimos anos estão revelando que as membranas de Golgi fornecem uma plataforma para a regulação de uma gama de processos celulares incluindo a polarização celular (Kupfer et al, 1983), migração dirigida (Millarte e Farhan, 2012), estresse (Sasaki e Yoshida, 2015), reparação do DNA (Farber-Katz et al., 2014), mitose (Rabouille e Kondylis, 2007), metabolismo (Abdel Rahman et al., 2015), respostas pró-inflamatórias (Chen e Chen, 2018) e autofagia (Yamamoto et al., 2012; Lamb et al., 2013). De fato, em células de mamíferos há agora evidências consideráveis de que o Golgi, como outras organelas intracelulares, pode atuar como um sensor de células (Farhan e Rabouille, 2011; Mayinger, 2011; Millarte e Farhan, 2012; Sasaki e Yoshida, 2015; Luini e Parashuraman, 2016; Gosavi e Gleeson, 2017; Makhoul et al., 2018). Também está ficando claro que a morfologia precisa do Golgi é relevante para a regulação de vários desses processos celulares (Makhoul et al., 2018). A associação da morfologia do Golgi com a sinalização foi confirmada a partir de uma tela de cinoma e fosfatoma que identificou uma grande coorte de quinases e fosfatase (20% do total no genoma) que influenciou a morfologia do Golgi (Chia et al., 2012). As mudanças na morfologia do Golgi incluíram ou a fragmentação do Golgi (perda da fita de Golgi) ou a formação de um Golgi muito compacto e condensado no local perinuclear. A relevância da actina na regulação da morfologia do Golgi foi destacada neste estudo pela identificação de várias kinases, por exemplo ROCK1 e PAK1, que regulam a dinâmica da actina e modulam a estrutura do Golgi (Chia et al., 2012). Outras análises do genoma também destacaram a probabilidade do Golgi poder receber e transmitir uma grande variedade de sinais que poderiam influenciar, não só as vias de transporte de membrana, mas também outros processos, apoptose, mitose, autofagia e respostas ao stress (Farhan et al., 2010; Millarte et al., 2015).
A estrutura da fita do Golgi é altamente dinâmica e pode sofrer uma remodelação muito rápida durante uma série de condições diferentes. Por exemplo, durante a mitose, a desmontagem da fita Golgi é um evento precoce na transição G2/M e desempenha um papel importante como ponto de controle do ciclo celular na promoção da entrada mitótica (Wei e Seemann, 2010; Corda et al., 2012). A regulação da dinâmica de Golgi é mediada por interações entre os andaimes moleculares localizados na membrana de Golgi e o citoesqueleto. A dinâmica MT pode regular a localização da fita de Golgi no centrossoma e o reposicionamento do Golgi para facilitar o tráfico polarizado e a secreção dirigida (Millarte e Farhan, 2012; Sanders e Kaverina, 2015). Além disso, componentes de membrana do Golgi também podem nuclear e estabilizar microtubulos tanto no cis- como no trans-Golgi, e portanto o próprio Golgi também é um MTOC (Efimov et al., 2007; Wu et al., 2016). Processos mediados por actina também contribuem para a forma e função do Golgi e pelo menos nove andaimes moleculares localizados no Golgi foram identificados que interagem com o citoesqueleto de actina. O aumento da polimerização da actina no Golgi resulta na dispersão da fita, enquanto que a inibição da polimerização da actina com drogas específicas como a latrunculina A resulta na compactação do Golgi (Lazaro-Dieguez et al., 2006; Makhoul et al., 2018, 2019). Dadas essas redes reguladoras que modulam a estrutura da fita de Golgi, consideramos muito plausível que o equilíbrio entre a fita de Golgi e as mini-fitas de Golgi possa definir as respostas qualitativas e quantitativas das vias de sinalização. A ligação entre a morfologia do Golgi e a sinalização também tem importantes ramificações na compreensão da base molecular de uma série de doenças que estão associadas à perda da fita de Golgi e ao aparecimento de um Golgi disperso. Por exemplo, a sobrevivência de algumas células cancerígenas tem se mostrado associada a um Golgi disperso, o que reduz o nível de apoptose (Farber-Katz et al., 2014; Petrosyan, 2015).
O que significa a fragmentação do Golgi?
O termo “fragmentação do Golgi” é comumente usado para descrever o estado morfológico de um Golgi disperso em células de mamíferos, como detectado por microscopia óptica corada com marcadores de Golgi. Um Golgi disperso é frequentemente observado em sistemas experimentais, por exemplo, quando as células são tratadas com drogas para perturbar o citoesqueleto, por exemplo, nocodazol (Wei e Seemann, 2010), quando o fluxo da membrana é perturbado, ou quando componentes de máquinas de transporte do Golgi, ou o sistema de interação citoesquelética, são derrubados, derrubados ou superexpressos (Zappa et al., 2018). Em vários estados fisiológicos, por exemplo, estresse (Serebrenik et al., 2018) e condições patológicas, particularmente câncer e neurodegeneração, o Golgi tem frequentemente perdido a típica localização juxtanuclear compacta e é observado por microscopia confocal como estruturas dispersas ao longo do citoplasma. Há um número crescente de relatos descrevendo a fragmentação do Golgi sob condições fisiológicas e patológicas (Figura 1A). Entretanto, um problema com o uso do termo “fragmentado” é que ele implica que a integridade estrutural do Golgi é perdida e que a morfologia associada à estrutura “fragmentada” representa uma organela desintegrada, anormal ou destruída. Em vários casos isto é claramente enganoso, pois as pilhas individuais do Golgi podem permanecer intactas e podem manter as funções clássicas da organela, nomeadamente a glicosilação e o transporte de membranas. De fato, mini-estacas de Golgi dispersas ocorrem em algumas células especializadas, como miooblastos diferenciados (Lu et al., 2001), neurônios diferenciados que contêm pilhas ou “postos avançados” individuais de Golgi ao longo dos dendritos (Lasiecka e Winckler, 2011), células parietais gástricas (Gunn et al., 2011) e células uroepiteliais da bexiga urinária (Kreft et al., 2010), sem deficiência aparente no transporte das membranas e na glicosilação. Ao contrário, a dispersão da fita de Golgi associada às condições experimentais e patológicas pode refletir uma mudança de equilíbrio dinâmico entre a morfologia compacta da fita e as mini-fitas individuais de Golgi ou pode resultar em perturbação da arquitetura da fita, bem como da integridade da pilha de Golgi. Esta é uma questão relevante uma vez que o estado morfológico preciso do Golgi irá muito provavelmente influenciar, em alguns casos, a eficiência do transporte e da glicosilação (Puthenveedu et al., 2006), e em outros casos uma variedade de redes de sinalização, mas não necessariamente o transporte ou a glicosilação. É importante diferenciar entre mini-pilhetas Golgi intactas e perda da integridade das pilhas Golgi na definição do que se entende por uma Golgi “fragmentada”. Aqui revisamos as diferentes morfologias do Golgi que foram detectadas e caracterizadas em cenários experimental, fisiológico e patológico.
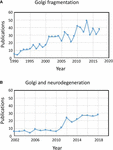
Figura 1. Publicações que identificam a fragmentação da fita Golgi. (A) Número de publicações por ano com o termo fragmentação da fita Golgi no título ou resumo. (B) Número de publicações por ano em que o Golgi foi examinado em doenças neurodegenerativas. Os dados são de Alexandru Dan Corlan. Medline trend: estatísticas anuais automatizadas dos resultados do PubMed para qualquer consulta, 2004. Recurso da Web na URL: http://dan.corlan.net/medline-trend.html. Acesso: 2019-04-29.
Rethinking the Terminology of Golgi Morphological States
As estruturas dos fragmentos de Golgi diferem dependendo da natureza do caminho envolvido para perturbar ou modular a fita de Golgi. É importante ter uma melhor caracterização ultra-estrutural dos “fragmentos” do Golgi após a perda da fita de Golgi, pois o resultado funcional é provavelmente muito diferente, dependendo das estruturas precisas do Golgi. Além das condições que resultam em uma fita Golgi alongada, podemos identificar na literatura pelo menos 4 cenários diferentes associados à “fragmentação” da fita Golgi. Estes são representados na Figura 2 e descritos da seguinte forma:
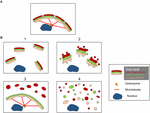
Figura 2. Modelo mostrando diferentes morfologias de Golgi após a “fragmentação” da estrutura da fita de Golgi. (A) Estrutura da fita Golgi intacta e (B) diferentes cenários mostrando a perda da fita Golgi. (1) Um cenário onde as mini-fitas de Golgi intactas estão dispersas pelo citoplasma; (2) Um cenário onde a integridade das fitas de Golgi dispersas está comprometida com cisterna encurtada, inchaço da cisterna e aumento das túbulas e vesículas associadas ao Golgi; (3) Um cenário onde há dispersão de um compartimento de Golgi. Aqui o TGN está selectivamente disperso pelo citoplasma enquanto o resto da pilha permanece numa estrutura de fitas; (4) Cenário onde há perda de fitas e pilhas com as membranas do Golgi dispersas predominantemente como túbulos e vesículas. Os números referem-se à classificação das morfologias de Golgi dada no texto.
(1) Conversão de fitas de Golgi em pilhas de Golgi. Aqui a perda (ou redução do comprimento) da fita de Golgi está associada a mini-fitas Golgi dispersas, intactas. Esta situação ocorre em vários tipos de células e também em sistemas modelo, tais como o tratamento com nocodazole ou a modulação dos níveis de TGN Golgi GCC88. O transporte por membrana parece em grande parte não ser afetado sob estas condições, com exceção de cargas possivelmente grandes (Ferraro et al., 2014; Lavieu et al., 2014), enquanto a sinalização mTOR é reduzida e a autofagia melhorada (Gosavi et al., 2018).
(2) Perda tanto da fita de Golgi quanto da integridade das pilhas de Golgi. Neste cenário, a estrutura da fita é perdida e, além disso, a cisterna das pilhas individuais é reduzida em comprimento e/ou número e também pode estar associada a compartimentos inchados, por exemplo, o knockout do GM130 (Liu et al., 2017) e mutações das subunidades COG (Blackburn e Lupashin, 2016). O transporte de membranas e a glicosilação podem ser afetados neste cenário juntamente com várias redes de sinalização.
(3) Dispersão de um compartimento Golgi. Neste cenário, apenas um compartimento de Golgi está disperso, como a dispersão do TGN recentemente reportada ativação do inflammasome NLRP3 (Chen e Chen, 2018). O restante da pilha/ribbon permanece inalterado. A identificação destas estruturas Golgi requer a incorporação de EM e microscopia óptica e um número de marcadores Golgi através da pilha para definir as mudanças precisas na morfologia do Golgi.
(4) Conversão da fita Golgi em elementos tubulovesiculares. Neste cenário, tanto a fita de Golgi quanto as pilhas estão amplamente perturbadas com aumentos dramáticos nas estruturas tubulovesiculares, como no caso da mitose (Wei e Seemann, 2017), vários medicamentos e tratamento de células com amilóide β (Joshi et al., 2014). Aqui se antecipa um impacto em muitas das funções do Golgi.
Não inferimos que a Figura 2 representa as únicas morfologias dos “fragmentos” do Golgi e é possível que cenários adicionais sejam identificados como as estruturas dos “fragmentos” do Golgi sejam investigados mais extensivamente.
Exemplos de uma Relação entre a Morfologia do Golgi e o Processo Celular
A coordenação das mudanças na morfologia do Golgi e vários processos celulares tem recebido considerável atenção. Para um resumo mais detalhado da informação de fundo dos processos influenciados pelas mudanças na morfologia do Golgi, tais como tráfico, glicosilação, stress, reparação do DNA, o leitor é referido a uma série de revisões recentes (Farhan e Rabouille, 2011; Millarte e Farhan, 2012; Sasaki e Yoshida, 2015; Gosavi e Gleeson, 2017; Makhoul et al., 2019). Abaixo estão alguns exemplos para destacar a gama de processos celulares que são regulados ou coordenados por diferentes estados morfológicos do Golgi. Em vários outros casos, tais como alguns cancros e respostas ao stress, a fita de Golgi está dispersa como fragmentos, contudo, a morfologia destes fragmentos de Golgi não tem sido bem caracterizada. A discussão aqui será focada nos exemplos onde a morfologia do Golgi é bem definida.
Reparo de DNA e Câncer
Existe uma associação íntima entre a morfologia do Golgi e a resposta do DNA aos danos (Farber-Katz et al., 2014). O cabo de membrana de Golgi, GOLPH3, é um oncogene e superexpressão de GOLPH3 resulta em maior sobrevivência celular após dano de DNA (Scott et al., 2009; Farber-Katz et al., 2014). Por outro lado, a perda de GOLPH3 previne a dispersão da fita de Golgi, melhora a fita de Golgi e promove a apoptose após a danificação do DNA. A resposta ao dano do DNA é mediada pela cinase DNA-PK, que fosforila o GOLPH3 e promove a “fragmentação” do Golgi ao melhorar a polimerização da actina nas membranas do Golgi (Dippold et al., 2009). mTOR é modulado pelas mudanças na morfologia do Golgi mediadas pelo GOLPH3 (Scott et al., 2009) e é provável que contribua para o resultado da resposta do DNA. Assim, a estrutura precisa do Golgi está ligada à sobrevivência celular e à apoptose. A identidade das estruturas do Golgi após a dispersão da fita de Golgi pelo fosfogolphth3 continua por ser caracterizada. Estudos futuros examinando a relação entre a fragmentação do Golgi e a sinalização de mTOR nas células cancerígenas serão claramente dignos de nota.
Desordens genéticas associadas à morfologia alterada do Golgi
Muitas doenças foram identificadas com desordens monogénicas causadas por mutações hereditárias de componentes associados a máquinas de transporte ou das enzimas residentes no Golgi. Muitas dessas doenças estão associadas à fragmentação da fita do Golgi, por exemplo, defeitos no complexo Golgi oligomérico conservado (COG) em doenças congênitas de glicosilação (Miller e Ungar, 2012). Em muitos casos, as doenças estão associadas a patologias restritas a um número limitado de órgãos ou tecidos. A base da especificidade dos tecidos é mal compreendida, mas provavelmente devido a deficiências na glicosilação e secreção e também a alterações nas redes de sinalização associadas à perda da fita de Golgi, como as respostas ao stress de Golgi. O Golgi em todas as linhas celulares de nocaute da subunidade COG apresentam alterações moderadas a severas na morfologia caracterizada pelo EM, associadas à perda da fita, cisterna dilatada e, em alguns casos, ruptura das mini-fitas. A aplicação do EM foi fundamental na definição das alterações morfológicas (Blackburn e Lupashin, 2016).
Neurodegenerativas
O estado do Golgi nas doenças neurodegenerativas tem recebido recentemente considerável atenção (Figura 1B). A perda da fita do Golgi é uma característica comum de muitas doenças neurodegenerativas, incluindo a doença de Alzheimer, Huntington, esclerose lateral amiotrófica e doença de Parkinson (Gonatas et al., 2006; Haase e Rabouille, 2015; Rabouille e Haase, 2015; Sundaramoorthy et al., 2015). É muito provável que as perturbações da arquitectura do Golgi nestas doenças contribuam para os processos patológicos. Na maioria dos casos, a estrutura morfológica precisa dos fragmentos do Golgi nestas doenças neurodegenerativas ainda não foi definida. No entanto, dois sistemas experimentais investigaram recentemente mudanças na estrutura do Golgi e degeneração neuronal com algum detalhe. Em primeiro lugar, foi demonstrado que a eliminação condicional do GM130, um golinho estrutural que regula a fita de Golgi, no sistema nervoso central, causa fragmentação de Golgi, atrofia de dendritos e degeneração neuronal em ratos (Liu et al., 2017). A análise EM das células de Purkinje GM130 KO mostrou uma redução do comprimento cisterna e empilhamento do Golgi e, além disso, os postos avançados de dendritos típicos de Golgi estavam ausentes nesses GM130 -/- células de Purkinje (Liu et al., 2017). Em um segundo estudo, neurônios hipocampais de ratos transgênicos para a mutação sueca da proteína precursora amilóide (APP) e uma subunidade de presenilina 1 mutante de γ-secretase, onde ambas as mutações estão associadas à doença de Alzheimer de início precoce, revelaram extensa fragmentação de Golgi por microscopia óptica (Joshi et al., 2014). O EM quantitativo de neurônios nesses ratos mostrou uma redução no número e comprimento das cisternas nas pilhas em comparação com neurônios de ratos do tipo selvagem. Além disso, as cisternas estavam inchadas. As mudanças na morfologia de Golgi nesses neurônios primários mostraram estar diretamente associadas com o elevado nível de produção de amilóideβ. Neurônios primários tratados com amilóide sintético-β também mostraram fragmentação semelhante do Golgi, assim como um aumento nas estruturas tubulovesiculares associadas à cisterna do Golgi em comparação com células não tratadas. A perda da fita de Golgi foi devida à fosforilação do GRASP65, uma proteína estrutural de Golgi que desempenha um papel fundamental na desmontagem da fita de Golgi e empilhamento em mitose (Joshi et al., 2014). A comparação destes dois estudos acima é informativa, pois os caminhos que mediaram as mudanças na morfologia do Golgi diferem em cada caso, levando a diferenças na morfologia dos “fragmentos” do Golgi. É necessário considerar como essas diferentes vias que afetam a morfologia do Golgi influenciam as respostas a jusante.
mTOR Signaling
Nosso laboratório estabeleceu uma abordagem experimental para perturbar o equilíbrio entre a fita do Golgi e as mini-fitas do Golgi, modulando a dose de GCC88, uma goma localizada no TGN. Esta estratégia permitiu estabelecer uma linha celular estável, HeLa-B6, que carece de uma fita de Golgi. Mostramos que GCC88 regula o equilíbrio entre fitas Golgi e mini-fitas por um processo dependente de actina (Makhoul et al., 2019) e identificou a intersectina-1 (ITSN1), um factor de troca de nucleótidos de guanina para cdc42 (Hussain et al., 2001), como um interagente de GCC88 responsável pela perda da fita Golgi (Makhoul et al., 2019). As análises das células de HeLa B6, que não possuem uma fita de Golgi, demonstraram uma redução da atividade do mTOR e um aumento associado da biogênese do autofagosoma (Gosavi et al., 2018). mTOR é uma das principais vias de sinalização das células eucarióticas e conhecida por ser um regulador negativo da autofagia (Wullschleger et al., 2006). Assim, o equilíbrio das pilhas de Golgi para a fita de Golgi tem um efeito direto sobre o caminho da mTORC1. O uso de marcadores específicos do compartimento cis e trans, e tomografia EM, foi crítico ao revelar as mudanças morfológicas na fita de Golgi.
Inflamação
Inflammasomas do sistema imunológico inato atuam como um andaime para ativação caspase 1-dependente de citocinas pró-inflamatórias (Broz e Dixit, 2016). O NLRP3 (domínio de ligação de nucleotídeos, família rica em leucina, domínio de pirina contendo-3) é um inflammasoma versátil que pode ser ativado por uma gama de estímulos microbianos e não-microbianos resultando na secreção de citocinas pró-inflamatórias interleucinas 1β (IL-1β) e interleucina 18 (IL-18) e morte celular programada por piroptose. Um estudo recente demonstrou que a ativação da NLRP3 citosólica por estímulos envolve o recrutamento de NLRP3 para membranas TGN dispersas para facilitar a montagem do andaime NLRP3 (Chen e Chen, 2018). A TGN dispersa, mas não os outros compartimentos subjacentes, recruta especificamente a NLRP3, via PI4P, para montar a ASC do complexo adaptador a jusante que sofre polimerização na região peri-nuclear antes de recrutar a caspase-1 para ativar a via de sinalização a jusante (Chen e Chen, 2018). Assim, esta importante descoberta demonstra que a modulação da arquitetura do TGN de forma seletiva, é crítica na ativação deste caminho. O uso de marcadores específicos do compartimento (TGN e cis-Golgi) juntamente com a análise óptica e EM foi crítico para revelar as alterações morfológicas na fita de Golgi.
Comentários Finais
A arquitectura precisa da morfologia de Golgi é definida pela microscopia óptica de alta resolução e pelo EM. A inclusão desta informação em futuros estudos no campo proporcionará uma visão considerável da dinâmica do Golgi, das vias de perturbação da estrutura da fita e das consequências funcionais associadas a estas diferentes vias.
Em resumo, propomos que o uso do termo “Golgi fragmentado” seja inadequado para descrever as estruturas do Golgi associadas a muitos tratamentos e condições e que as diferenças nestas estruturas do Golgi sejam fisiologicamente relevantes. Dada a natureza dinâmica do aparelho de Golgi, é possível que haja um equilíbrio entre os diferentes estados morfológicos do Golgi a qualquer momento, ou seja, mini-pilotos e estruturas de fitas. O entendimento do equilíbrio dinâmico entre as diferentes morfologias do Golgi em detalhe molecular é crítico para uma apreciação completa desta organela durante os processos celulares normais e também sob condições patológicas. Será fascinante ver o que se desdobra à medida que aprendemos mais sobre as funções de detecção celular desta organela complexa e a relação entre estas funções e as estruturas de Golgi.
Data Availability
Todos os conjuntos de dados analisados para este estudo estão incluídos no manuscrito e nos arquivos suplementares.
Todos os autores listados fizeram uma contribuição substancial, direta e intelectual ao trabalho e o aprovaram para publicação.
Funding
Este trabalho foi apoiado por financiamento do Conselho Australiano de Pesquisa (DP160102394).
Conflict of Interest Statement
Os autores declaram que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
A abreviações
GM130, proteína de matriz cis-Golgi; GMAP210, proteína associada ao microtubo de Golgi; GOLPH3, fosfoproteína 3 de Golgi; GRASP, proteína de remontagem de Golgi; MTOC, centro organizador do microtubo; mTOR, alvo mecanicista da rapamicina; PI4P, fosfatidilinositol-4-fosfato; TGN, rede trans-Golgi.
Abdel Rahman, A. M., Ryczko, M., Nakano, M., Pawling, J., Rodrigues, T., Johswich, A., et al. (2015). Golgi N-glycan branching N-acetylglucosaminyltransferases I, V e VI promovem a absorção de nutrientes e o metabolismo. Glycobiology 25, 225-240. doi: 10.1093/glycob/cwu105
PubMed Abstract | CrossRef Full Text | Google Scholar
Blackburn, J. B., e Lupashin, V. V. (2016). Criação de knockouts de subunidades conservadas do complexo oligomérico golgi usando a edição genética mediada por CRISPR pareada com uma estratégia de seleção baseada em defeitos de glicosilação associados à função do complexo COG deficiente. Métodos Mol. Biol. 1496, 145-161. doi: 10.1007/978-1-4939-6463-5_12
PubMed Abstract | CrossRef Full Text | Google Scholar
Boncompain, G., e Perez, F. (2013). As muitas rotas do tráfico dependente do Golgi. Histochem. Cell Biol. 140, 251-260. doi: 10.1007/s00418-013-1124-7
PubMed Abstract | CrossRef Full Text | Google Scholar
Broz, P., e Dixit, V. M. (2016). Inflammasomes: mecanismo de montagem, regulação e sinalização. Nat. Rev. Immunol. 16, 407-420. doi: 10.1038/nri.2016.58
PubMed Abstract | CrossRef Full Text | Google Scholar
Chen, J., and Chen, Z. J. (2018). PtdIns4P em rede trans-Golgi dispersa medeia a ativação do inflammasome NLRP3. Nature 564, 71-76. doi: 10.1038/s41586-018-0761-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Chia, J., Goh, G., Racine, V., Ng, S., Kumar, P., and Bard, F. (2012). O rastreio RNAi revela uma grande rede de sinalização que controla o aparelho Golgi em células humanas. Mol. Syst. Biol. 8:629. doi: 10.1038/msb.2012.59
PubMed Abstract | CrossRef Full Text | Google Scholar
Corda, D., Barretta, M. L., Cervigni, R. I., e Colanzi, A. (2012). Fragmentação do complexo Golgi na transição G2/M: um ponto de controle do ciclo celular baseado em organelas. IUBMB Life 64, 661-670. doi: 10.1002/iub.1054
PubMed Abstract | CrossRef Full Text | Google Scholar
De Matteis, M. A., Mironov, A. A., e Beznoussenko, G. V. (2008). A Fita Golgi e a Função dos Golgins. Nova Iorque, NY: Springer-Verlag/Wein.
Google Scholar
Dippold, H. C., Ng, M. M., Farber-Katz, S. E., Lee, S. K., Kerr, M. L., Peterman, M. C., et al. (2009). GOLPH3 faz a ponte fosfatidilinositol-4- fosfato e actomiosina para esticar e moldar o Golgi para promover o florescimento. Cell 139, 337-351. doi: 10.1016/j.cell.2009.07.052
PubMed Abstract | CrossRef Full Text | Google Scholar
Efimov, A., Kharitonov, A., Efimova, N., Loncarek, J., Miller, P. M., Andreyeva, N., et al. (2007). Asymmetric CLASP-dependent nucleation of noncentrosomal microtubules at the trans-Golgi network. Dev. Cell 12, 917-930. doi: 10.1016/j.devcel.2007.04.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Farber-Katz, S. E., Dippold, H. C., Buschman, M. D., Peterman, M. C., Xing, M., Noakes, C. J., et al. (2014). Os danos no DNA provocam a dispersão de Golgi via DNA-PK e GOLPH3. Cell 156, 413-427. doi: 10.1016/j.cell.2013.12.023
PubMed Abstract | CrossRef Full Text | Google Scholar
Farhan, H., e Rabouille, C. (2011). Sinalização de e para o caminho do secretariado. J. Cell Sci. 124, 171-180. doi: 10.1242/jcs.076455
PubMed Abstract | CrossRef Full Text | Google Scholar
Farhan, H., Wendeler, M. W., Mitrovic, S., Fava, E., Silberberg, Y., Sharan, R., et al. (2010). MAPK sinalizando para o caminho inicial do secretariado revelado pela triagem funcional kinase/fosfátase. J. Cell Biol. 189, 997-1011. doi: 10.1083/jcb.200912082
PubMed Abstract | CrossRef Full Text | Google Scholar
Ferraro, F., Kriston-Vizi, J., Metcalf, D. J., Martin-Martin, B., Freeman, J., Burden, J. J., et al. (2014). Um controle a dois níveis, baseado em Golgi, do tamanho das organelas sustenta a plasticidade funcional das células endoteliais. Dev. Cell 29, 292-304. doi: 10.1016/j.devcel.2014.03.021
PubMed Abstract | CrossRef Full Text | Google Scholar
Gonatas, N. K., Stieber, A., and Gonatas, J. O. (2006). Fragmentação do aparelho de Golgi em doenças neurodegenerativas e morte celular. J. Neurol. Sci. 246, 21-30. doi: 10.1016/j.jns.2006.01.019
PubMed Abstract | CrossRef Full Text | Google Scholar
Gosavi, P., e Gleeson, P. A. (2017). A função da estrutura da fita Golgi – Um mito duradouro se desdobra! Bioessays 39:1700063. doi: 10.1002/bies.201700063
PubMed Abstract | CrossRef Full Text | Google Scholar
Gosavi, P., Houghton, F. J., Mcmillan, P. J., Hanssen, E., e Gleeson, P. A. (2018). A fita de Golgi em células de mamíferos regula negativamente a autofagia através da modulação da atividade mTOR. J. Cell Sci. 131:jcs211987. doi: 10.1242/jcs.211987
PubMed Abstract | CrossRef Full Text | Google Scholar
Gunn, P. A., Gliddon, B. L., Londrigan, S. L., Lew, A. M., Van Driel, I. R., e Gleeson, P. A. (2011). O aparato de Golgi nas células parietais gástricas endomembranas existem como mini-estacas funcionais estáveis dispersas por todo o citoplasma. Biol. Cell 103, 559-572. doi: 10.1042/BC20110074
PubMed Abstract | CrossRef Full Text | Google Scholar
Haase, G., e Rabouille, C. (2015). Fragmentação do Golgi em neurônios motores ALS. Novos mecanismos visando microtubos, amarras, e vesículas de transporte. Frente. Neurosci. 9:448. doi: 10.3389/fnins.2015.00448
PubMed Abstract | CrossRef Full Text | Google Scholar
Hussain, N. K., Jenna, S., Glogauer, M., Quinn, C. C., Wasiak, S., Guipponi, M., et al. (2001). A proteína endocítica intersetina-l regula a montagem da actina via Cdc42 e N-WASP. Nat. Cell Biol. 3, 927-932. doi: 10.1038/ncb1001-927
PubMed Abstract | CrossRef Full Text | Google Scholar
Joshi, G., Chi, Y., Huang, Z., e Wang, Y. (2014). A fragmentação induzida pela Abeta na doença de Alzheimer aumenta a produção de Abeta. Proc. Natl. Acad. Sci. U.S.A. 111, E1230-E1239. doi: 10.1073/pnas.1320192111
PubMed Abstract | CrossRef Full Text | Google Scholar
Kreft, M. E., Di Giandomenico, D., Beznoussenko, G. V., Resnik, N., Mironov, A. A., e Jezernik, K. (2010). Fragmentação do aparelho de Golgi como mecanismo responsável pela entrega uniforme de uroplakins à membrana plasmática apical das células uroepiteliais. Biol. Cell 102, 593-607. doi: 10.1042/bc20100024
PubMed Abstract | CrossRef Full Text | Google Scholar
Kupfer, A., Dennert, G., and Singer, S. J. (1983). Polarização do aparelho de Golgi e do centro organizador do microtubo dentro de células assassinas naturais clonadas ligadas aos seus alvos. Proc. Natl. Acad. Sci. U.S.A. 80, 7224-7228. doi: 10.1073/pnas.80.23.7224
PubMed Abstract | CrossRef Full Text | Google Scholar
Lamb, C. A., Yoshimori, T., and Tooze, S. A. (2013). O autofagosoma: origens desconhecidas, complexo de biogénese. Nat. Rev. Mol. Cell Biol. 14, 759-774. doi: 10.1038/nrm3696
PubMed Abstract | CrossRef Full Text | Google Scholar
Lasiecka, Z. M., e Winckler, B. (2011). Mecanismos do tráfico de membranas polarizadas em neurônios – focalizando em endossomos. Mol. Cell. Neurociências. 48, 278-287. doi: 10.1016/j.mcn.2011.06.013
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavieu, G., Dunlop, M. H., Lerich, A., Zheng, H., Bottanelli, F., e Rothman, J. E. (2014). A estrutura de fitas Golgi facilita o transporte anterógrado de grandes cargas. Mol. Biol. Cell 25, 3028-3036. doi: 10.1091/mbc.E14-04-093
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazaro-Dieguez, F., Jimenez, N., Barth, H., Koster, A. J., Renau-Piqueras, J., Llopis, J. L., et al. (2006). Os filamentos de actina estão envolvidos na manutenção da morfologia de Golgi cisternae e do pH intra-Golgi. Motil celular. Cytoskeleton 63, 778-791. doi: 10.1002/cm.20161
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, C., Mei, M., Li, Q., Roboti, P., Pang, Q., Ying, Z., et al. (2017). A perda da margem de ouro GM130 causa perturbações no Golgi, perda dos neurónios Purkinje e ataxia em ratos. Proc. Natl. Acad. Sci. U.S.A. 114, 346-351. doi: 10.1073/pnas.1608576114
PubMed Abstract | CrossRef Full Text | Google Scholar
Lu, Z., Joseph, D., Bugnard, E., Zaal, K. J., and Ralston, E. (2001). Reorganização do complexo Golgi durante a diferenciação muscular: visualização em células vivas e mecanismo. Mol. Biol. Cell 12, 795-808. doi: 10.1091/mbc.12.4.795
PubMed Abstract | CrossRef Full Text | Google Scholar
Luini, A., and Parashuraman, S. (2016). Sinalização no Golgi: detecção e controle dos fluxos de membrana. Moeda. Opinião. Cell Biol. 39, 37-42. doi: 10.1016/j.ceb.2016.01.014
PubMed Abstract | CrossRef Full Text | Google Scholar
Makhoul, C., Gosavi, P., Duffield, R., Delbridge, B., Williamson, N. A., e Gleeson, P. A. (2019). Intersectin-1 interage com o GCC88 de ouro para unir a rede actin e a arquitectura Golgi. Mol. Biol. Cell 30, 370-386. doi: 10.1091/mbc.E18-05-0313
PubMed Abstract | CrossRef Full Text | Google Scholar
Makhoul, C., Gosavi, P., e Gleeson, P. A. (2018). A arquitetura Golgi e o sensoriamento celular. Biochem. Soc. Trans. 46, 1063-1072. doi: 10.1042/BST20180323
PubMed Abstract | CrossRef Full Text | Google Scholar
Mayinger, P. (2011). Sinalização no Golgi. Cold Spring Harb. Perspicácia. Biol. 3:a005314.
Google Scholar
Millarte, V., Boncompain, G., Tillmann, K., Perez, F., Sztul, E., e Farhan, H. (2015). Phospholipase C gamma1 regula o tráfico de secretariado precoce e a migração celular através da interação com a p115. Mol. Biol. Cell 26, 2263-2278. doi: 10.1091/mbc.E15-03-0178
PubMed Abstract | CrossRef Full Text | Google Scholar
Millarte, V., e Farhan, H. (2012). O Golgi na migração celular: regulação por transdução de sinal e suas implicações na metástase celular cancerígena. ScientificWorldJournal 2012:498278. doi: 10.1100/2012/498278
PubMed Abstract | CrossRef Full Text | Google Scholar
Miller, V. J., e Ungar, D. (2012). Re’COG’nition no Golgi. Traffic 13, 891-897. doi: 10.1111/j.1600-0854.2012.01338.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Petrosyan, A. (2015). Onco-Golgi: a fragmentação é uma porta para a progressão do câncer? Biochem. Mol. Biol. J. 1:16.
PubMed Abstract | Google Scholar
Puthenveedu, M. A., Bachert, C., Puri, S., Lanni, F., e Linstedt, A. D. (2006). A fusão cisterna lateral dependente de GM130 e GRASP65 permite a distribuição uniforme da enzima Golgi. Nat. Cell Biol. 8, 238-248. doi: 10.1038/ncb1366
PubMed Abstract | CrossRef Full Text | Google Scholar
Rabouille, C., and Haase, G. (2015). Editorial: golgi patology in neurodegenerative diseases. Frente. Neurosci. 9:489. doi: 10.3389/fnins.2015.00489
PubMed Abstract | CrossRef Full Text | Google Scholar
Rabouille, C., and Kondylis, V. (2007). Golgi ribbon unlinking: um ponto de verificação G2/M baseado em organelas. Cell Cycle 6, 2723-2729. doi: 10.4161/cc.6.22.4896
PubMed Abstract | CrossRef Full Text | Google Scholar
Rambourg, A., and Clermont, Y. (1997). Estrutura Tridimensional do Aparelho de Golgi em Células de Mamíferos. Basiléia: Birkhauser.
Google Scholar
Sanders, A. A., e Kaverina, I. (2015). Nucleação e dinâmica de microtubos derivados de Golgi. Frente. Neurosci. 9:431. doi: 10.3389/fnins.2015.00431
CrossRef Full Text | Google Scholar
Sasaki, K., e Yoshida, H. (2015). Respostas de auto-regulação-estresse Organelle no ER, Golgi, mitocôndria e lisossoma. J. Biochem. 157, 185-195. doi: 10.1093/jb/mvv010
PubMed Abstract | CrossRef Full Text | Google Scholar
Scott, K. L., Kabbarah, O., Liang, M. C., Ivanova, E., Anagnostou, V., Wu, J., et al. (2009). GOLPH3 modula a sinalização de mTOR e a sensibilidade à rapamicina no câncer. Nature 459, 1085-1090. doi: 10.1038/nature08109
PubMed Abstract | CrossRef Full Text | Google Scholar
Serebrenik, Y. V., Hellerschmied, D., Toure, M., Lopez-Giraldez, F., Brookner, D., and Crews, C. M. (2018). O desdobramento da proteína-alvo revela uma resposta ao stress transcripcional específica de Golgi. Mol. Biol. Cell 29, 1284-1298. doi: 10.1091/mbc.E17-11-0693
PubMed Abstract | CrossRef Full Text | Google Scholar
Sundaramoorthy, V., Sultana, J. M., e Atkin, J. D. (2015). Fragmentação do Golgi na esclerose lateral amiotrófica, uma visão geral dos possíveis gatilhos e consequências. Frente. Neurosci. 9:400. doi: 10.3389/fnins.2015.00400
PubMed Abstract | CrossRef Full Text | Google Scholar
Wei, J. H., e Seemann, J. (2010). Desembrulhando a fita Golgi. Traffic 11, 1391-1400. doi: 10.1111/j.1600-0854.2010.01114.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wei, J. H., e Seemann, J. (2017). Desmontagem da fita Golgi durante a mitose, diferenciação e progressão da doença. Moeda. Opinião. Cell Biol. 47, 43-51. doi: 10.1016/j.ceb.2017.03.008
PubMed Abstract | CrossRef Full Text | Google Scholar
Wu, J., De Heus, C., Liu, Q., Bouchet, B. P., Noordstra, I., Jiang, K., et al. (2016). Caminho molecular de organização dos microtubos no aparelho de Golgi. Dev. Cell 39, 44-60. doi: 10.1016/j.devcel.2016.08.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Wullschleger, S., Loewith, R., and Hall, M. N. (2006). Sinalização TOR em crescimento e metabolismo. Cell 124, 471-484.
Google Scholar
Yamamoto, H., Kakuta, S., Watanabe, T. M., Kitamura, A., Sekito, T., Kondo-Kakuta, C., et al. (2012). Atg9 vesículas são uma importante fonte de membranas durante os primeiros passos da formação do auto-fagosoma. J. Cell Biol. 198, 219-233. doi: 10.1083/jcb.201202061
PubMed Abstract | CrossRef Full Text | Google Scholar
Zappa, F., Failli, M., and De Matteis, M. A. (2018). O complexo Golgi na doença e na terapia. Moeda. Opin. Cell Biol. 50, 102-116. doi: 10.1016/j.ceb.2018.03.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Leave a Reply